
Выпуск 279
Лаборатория Наномир
Когда реальность открывает тайны,
уходят в тень и
меркнут чудеса ...
Готовимся к участию в конкурсе CASP10
diprospan: Почему один и тот же пост в среду 11 янв 2012 в 12:02 выглядит в оригинале eng и после перевода rus по разному ?
Kushelev: Это ещё мелочи. Почему утром в теме было 4 страницы, а вечером осталась одна? Похоже, что в конкурсе CASP нас будут ждать если не тройные стандарты, то двойные стандарты точно ...
Напомню, что в конкурсе CASP3 перед розыгрышем призов из списка участников неожиданно исчезло 5 лабораторий, в т.ч. и лаборатория Наномир: http://nanoworld88.narod.ru/forum/casp/cafasp.htm
Руководитель конкурса сообщил, что данные, накопленные на сервере CAFASP за полтора месяца, были удалены "по техническим причинам".
Вероятно, по тем же причинам в начале февраля исчезли все мои сообщения на форуме FORCASP в теме, которую я открыл 11 декабря 2011 года. Но, рукописи не горят, поэтому желающие могут прочесть копию уничтоженной темы: http://nanoworld88.narod.ru/forum/casp/index.htm
Кушелев: Уважаемая Виктория!
Вам удалось зарегистрироваться на конкурсе CASP ?
Если да, то расскажите, как это делается. Я по-английски практически не понимаю, поэтому без Вашей помощи не смогу зарегистрировать лабораторию Наномир...
Судя по тому, как они отредактировали мою тему на форуме FORCASP, нас ждёт конкурс если не тройных, то двойных стандартов как минимум ![]()
Оригинал темы выглядел так: http://nanoworld88.narod.ru/forum/casp/index.htm
Victoria: Я вчера ( 2012-02-06 ) зарегистрировалась на сайте CASP как персона, а не как организация или сервер, и уже получила подтверждение. Ещё я поинтересовалась, как они будут рассылать вводную информацию и в каком формате хотели бы получать результаты. Сейчас жду ответ с объяснениями.С регистрацией ничего сложного: заходите на их сайт:http://www.predictioncenter.org/, справа в Menu в перечне выбираете PC Registration, вываливается страничка для регистрации он-лайн, в которой Вы заполняете окошечки, отмеченные красной звёздочкой. После заполнения их на английском языке нажимаете submit (подтверждаю) и всё. Будут проблемы, спрашивайте.
Kushelev: Огромное спасибо!

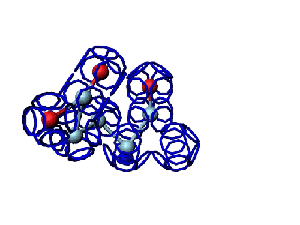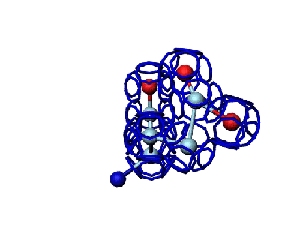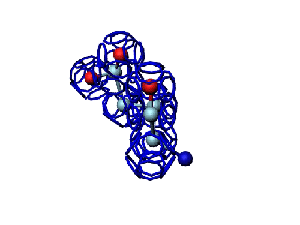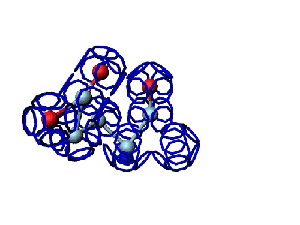
Victoria: Вы не заметили, что на этой модели формирования пептидной связи между n и n+1 аминокислотами представлены задом наперёд?
Я имею в виду, что первую аминокислоту нужно развернуть зеркально (N должен быть правее СА) и к её СА атому присоединить не СА+1 атом, а N+1 атом п+1 аминокислоты и только потом к нему СА+1.
Кушелев: Заметил конечно.
А в программе Дениса Савина этой ошибки вроде уже нет. Это модель Met-Ala (первый метионин, второй аланин). Вроде правильно. Да?
Victoria: Пока только фотки с пояснениями. Если что-либо плохо видно, то уточняйте. Могу гарантировать соответствие основным характеристикам альфа-спирали: водородные связи формируются между пептидными группами 1-го и 4-го аминокислотных остатков, на виток -- 4, 5 а.о., СО-группы смотрят вверх, радикалы вниз, закручивается вправо. В общем, такая спираль мне нравится
А это модель рибозы с углеродами-тетраэдрами:
Кушелев: Грандиозно!


Скрипт: element = Hedra family:0 scalep:100 scaleq:100 scaler:100 radius:1 pos:[0,0,0] p:0.4; Converttomesh element

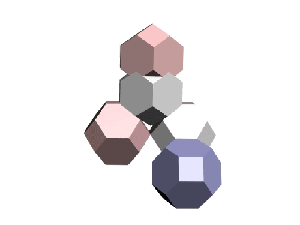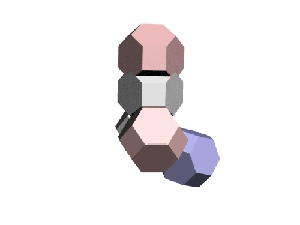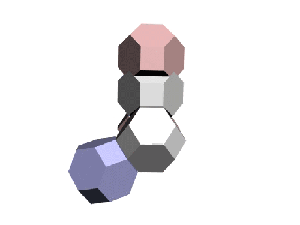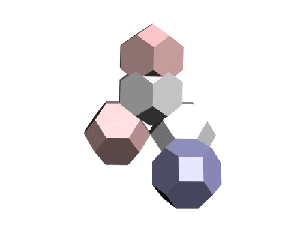
Скрипт:
element1 = Hedra family:1 scalep:100 scaleq:100 scaler:100 radius:1 pos:[0,0,0] p:0.4 wirecolor:[250,250,250]; Converttomesh element1
element2 = copy element1 wirecolor:[255,200,200]
move element2 [0,0,1]
-- attach element element2
element3 = copy element1 wirecolor:[255,200,200]
rotate element3 90 [0,0,1]
rotate element3 90 [1,0,0]
move element3 [0.95,0,-1.2]
element4 = copy element3 wirecolor:[250,250,250]
move element4 [-1.9,0,0]
element5 = copy element4 wirecolor:[190,190,255]
move element5 [0,1.3,-0.9]
rotate element5 90 [0,0,1]
rotate element5 110 [1,0,0]
g1 = group #(element1,element2,element3,element4,element5)
g1.pivot = [0,0,0]
g1.rotation.controller[3].controller.value = 0
animate on
at time 100 g1.rotation.controller[3].controller.value = 360
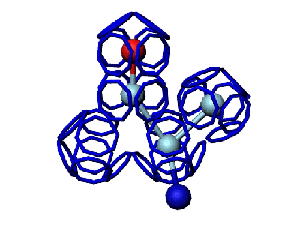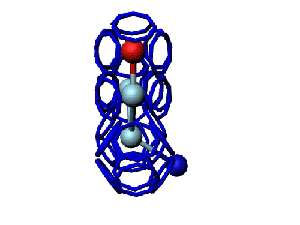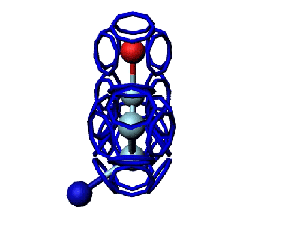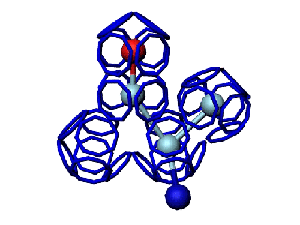
Исходный файл dne:
60FT
FT source 1..3
FT CDS <1..3
XX
SQ Sequence 250 BP; 36 A; 105 C; 78 G; 31 T; 0 other;
gcc
//
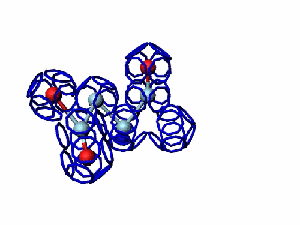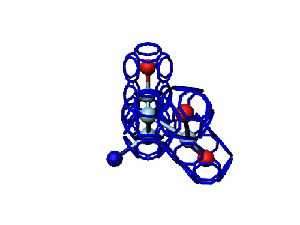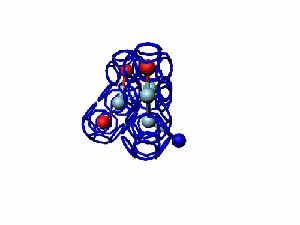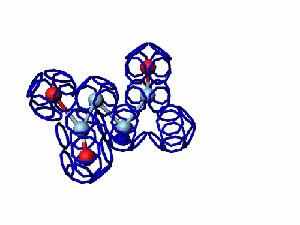
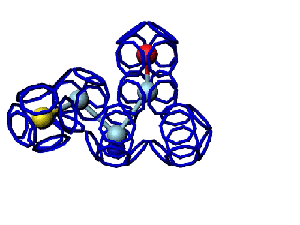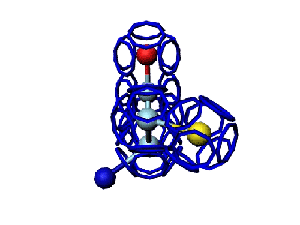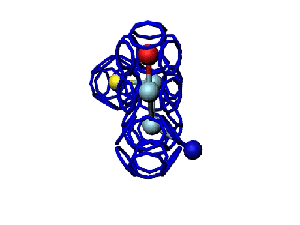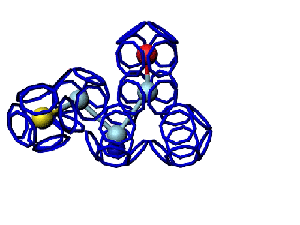
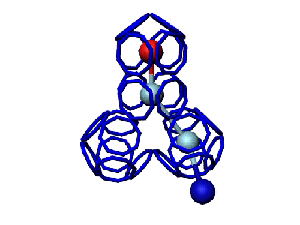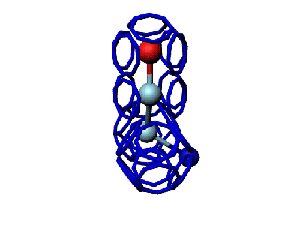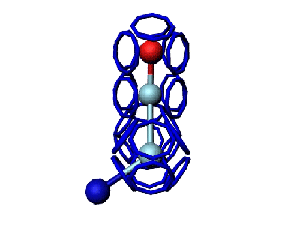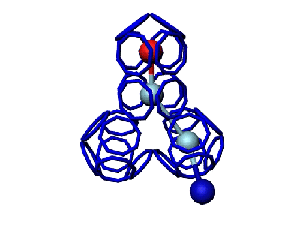
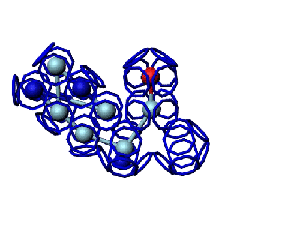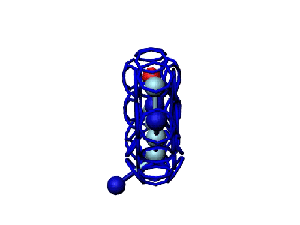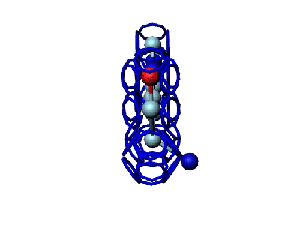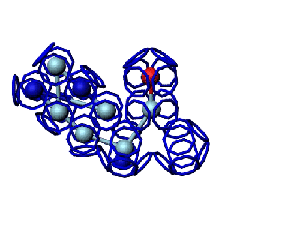
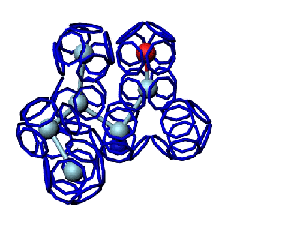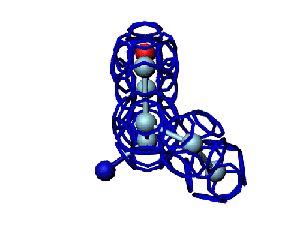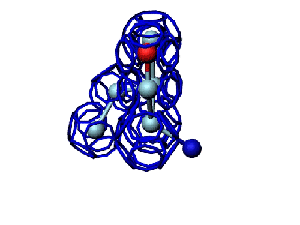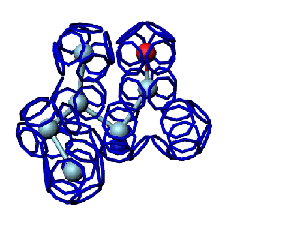
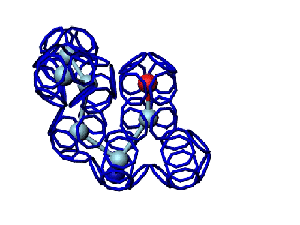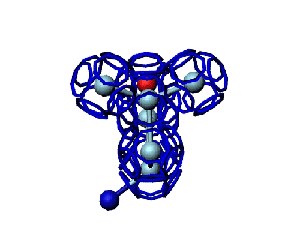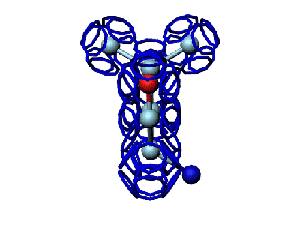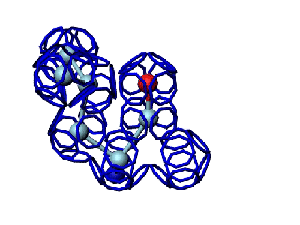
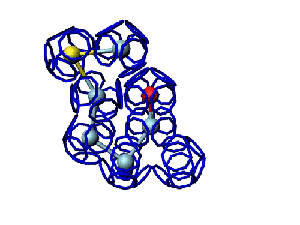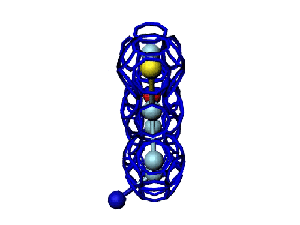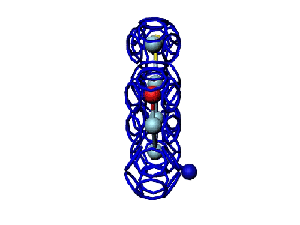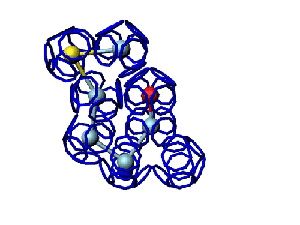
Модель пролина не реализована ... ( Pro/P по коду ccc )
Модель серина-lamda не реализована ... ( Ser_lamda/S по коду tcc )
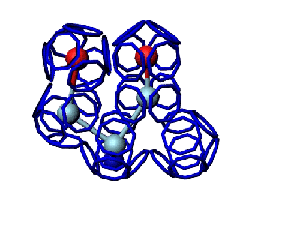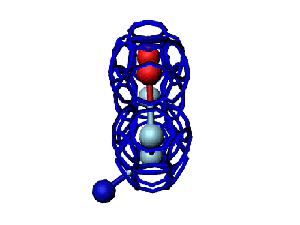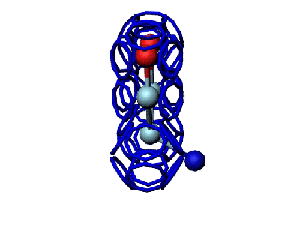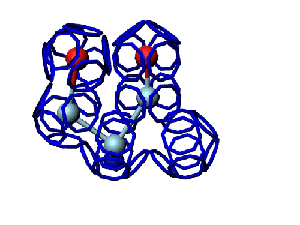
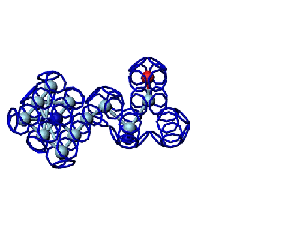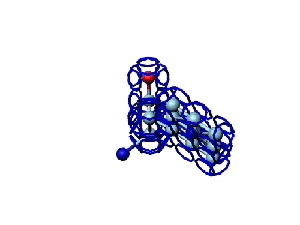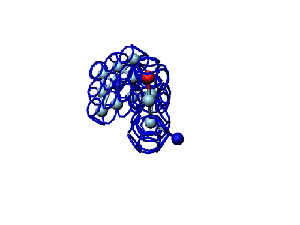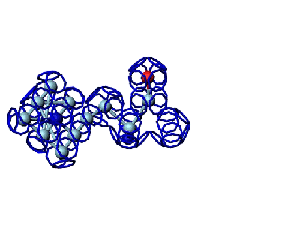
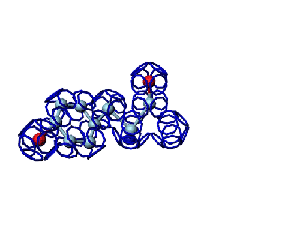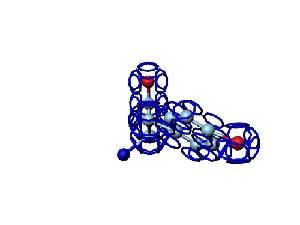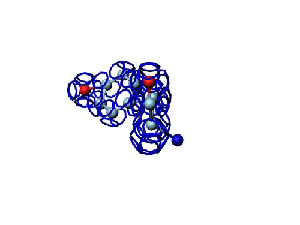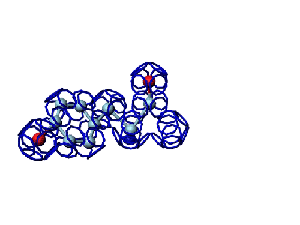
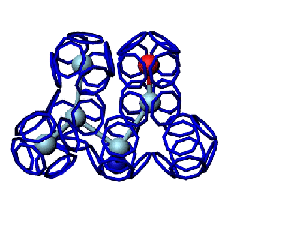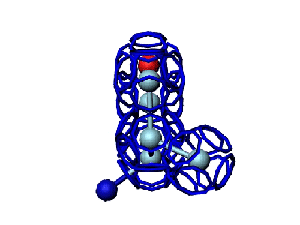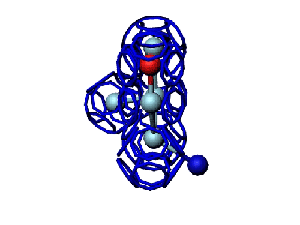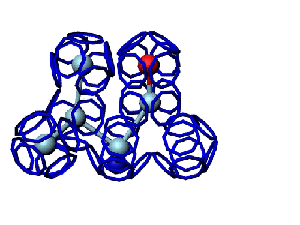
Кушелев: Вообще-то Фотошопу без разницы, сколько аминокислот нужно отразить в зеркале. Для меня существовала другая проблема, как преобразовать pdb-файл. Оказалось, что достаточно поменять знаки в одном из столбцов координат, например, во втором (Y-координаты всех атомов). С этой задачей (а также с задачей масштабирования) легко справляется скрипт для PyMOL. Но это - временная мера. На днях будет завершен первый этап модернизации скрипта Савина-Кушелева, после чего он будет сохранять pdb-файл (промасштабированный и зеркально преобразованный). На втором этапе модернизации будут исправлены ошибки и недоделки предыдущих версий. В частности, будет доделана модель пролина и исправлена модель серина-lamda.
Кушелев: Электроны атомов, связанных ковалентно, образуют общую молекулярную поверхность.


Подробнее это объясняется здесь: http://www.nanoworld.org.ru/data/01/data/images/slides/990410/index.htm
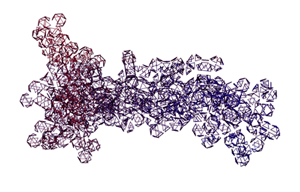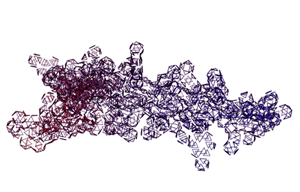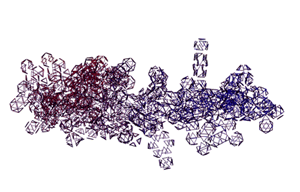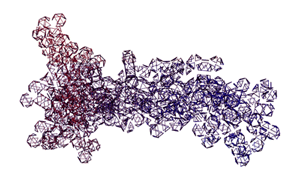
Кушелев: А куда же она денется, если многогранная модель - это упрощение кольцегранной:
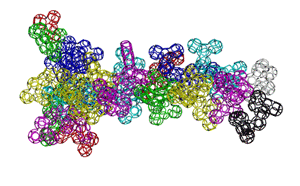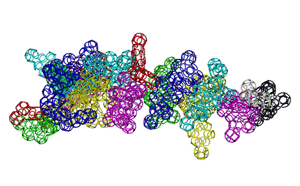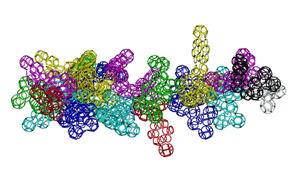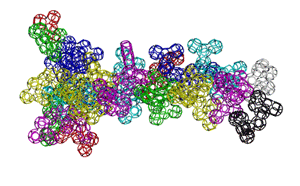
Подробнее: http://nanoworld88.narod.ru/data/241.htm

Подробнее: http://nanoworld88.narod.ru/forum/casp/001.htm
Victoria: Сегодня (2012-02-08), я получила ответ от Андрея Кристафовича (конкурс CASP), что они больше не делают предсказания вторичной структуры в CASP.Поэтому, до тех пор пока не будет реализован в моей программе файл PDB, я -- вне этого конкурса ![]() .
.
Kushelev: А какие конкурсы существуют сегодня?
Если существует один конкурс определения третичной структуры белка, то мы можем вместе в нём участвовать. Я могу отвечать за подготовку pdb-файлов, а Вы, например, можете находить dne или fasta-файлы по аминокислотной последовательности и вести переписку с организаторами конкурса на английском языке. В любом
случае Ваш алгоритм так слабо отличается от моего, что экспериментальные методы этого различия не заметят ![]()
Мне по почте пришло письмо от CASP10:
Цитата: We have released seventh CASP ROLL target today and are planning on releasing another one later this week. There are also good prospects that we will have one or two more targets for the next week release. Please also be reminded that target R01 expires in two days.
Конец цитаты.
Помогите перевести на русский...
Victoria: Перевод: Сегодня мы выпустили седьмую цель CASP ROLL и планируем выпуск остальных позже на этой неделе. Также имеются хорошие перспективы, что у нас будут одна или еще две цели для следующего недельного выпуска. Пожалуйста, напомните, что цель R01 истекает через два дня.
Кушелев: Понятно. А где эту цель R01 можно увидеть?
Кушелев: Стандартные вьюеры показывают модели по координатам из файла в стандарте PDB. Поэтому этот файл придётся сохранять из программы TSP. А это может сделать только программист. Мой программист, который занимается модификацией скрипта Дениса Савина, вряд ли сможет это сделать быстро, т.к. со скриптом он разбирается уже около 10 дней и пока непонятно, сколько дней ещё понадобится. А нам первые данные, как я понял, нужно отправить в крайнем случае завтра. Спасти может только Prosolver, который знает свою программу. И только в том случае, если он быстро разберётся, как сохранять файл в стандарте PDB. Есть ещё вариант, но он требует денег, которых у нас с Вами сегодня нет...
Поэтому я бы начал сегодня совместное (с Вами) участие в CASP10, а параллельно можно пытаться решать проблему сохранения данных из программы TSP. Получится, хорошо, не получится, значит хотя бы один вариант pdb-файлов мы отправим на конкурс. Вы зря переживаете, т.к. на конкурсе вряд ли заметят различия между программами Пикотех и TSP ![]()
А Вы поняли, где находится "цель R01", которую нужно отработать до завтра?
Victoria: Понятия не имею. Попросите их прислать, а заодно и нуклеотидную последовательность.
Kushelev: Я не в курсе, кого просить и как? Я же английский не знаю ![]() Может быть Вы сможете?
Может быть Вы сможете?
Умные люди советуют нам с Вами объединить усилия в этом конкурсе. Вместе у нас будет больше шансов дойти до финиша, чем по отдельности ![]()
Вы же можете сразу уточнить, что у Вас есть другой алгоритм, который Вы считаете более точным. Как только он будет реализован, Вы сможете продемонстрировать его возможности. И для этого не обязательно ждать ещё 2 года до следующего конкурса CASP ![]()
Я вроде бы нашёл список входных файлов CASP10: http://predictioncenter.org/casprol/targetlist.cgi
Это он?
Я правильно понял, что нужно зарегистрироваться не только на сайте CASP, но и здесь: http://www.predictioncenter.org/casprol/registration.cgi
Kushelev (2012-02-08 12:21): Я так понял, что нам нужно в первую очередь получить pdb-файл по этой аминокислотной последовательности: http://predictioncenter.org/casprol/target.cgi?id=11&view=all
Это оно?
http://www.ebi.ac.uk/ena/data/view/CAA24435&display=text
Получить структуру белка оказалось проще, чем зарегистрироваться на конкурсе CASP ![]()
Естественно, что дальний порядок скорее всего другой, т.е. мы видим то, что сошло с рибосомы и дальше может сложиться в более компактную глобулу, но тут уже нужно включать физико-химическое моделирование...
В этом нам помогут стандартные браузеры pdb-файлов, например, Swiss Viewer. А промасштабировать и зеркально преобразовать нам поможет скрипт PyMOL...
pdb-файл открыт в программе PyMOL
Промасштабирован и зеркально преобразован.
Преобразованный pdb-файл открыт в программе Swiss Viewer
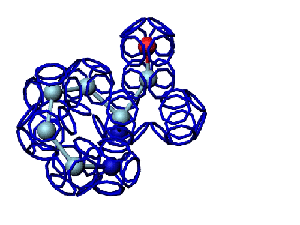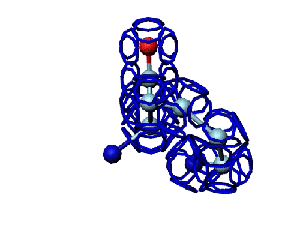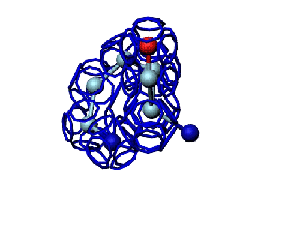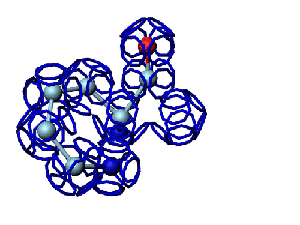
Программа PyMOL показывает часть структуры белка в виде спирали.
Сферами программа PyMOL тоже показывает только часть белка...
На этом фрагменте белка обнаруживается некая характерная форма, которая, вероятно, имеет функциональное назначение...
Ну что же, теперь нужно успеть отправить полученный с помощью программы Пикотех pdb-файл на конкурс CASP10 :) Виктория сообщила, что крайний срок - завтра. Как бы не опоздать...
Если не удаётся выйти на форум (он последнюю неделю подвержен ддос-атакам), можно посмотреть сохранённую копию: http://nanoworld88.narod.ru/forum/index.htm
Для срочной связи можно воспользоваться скайпом ( kushelev2009 ) и телефонами: +7-926-5101703, +7-903-2003424, +7-916-8265031. Будем рады Вашему участию в конкурсе CASP10 и в других мероприятиях лаборатории Наномир.
В настоящее время нашей задачей является отправить на конкурс CASP10 первый PDB-файл: http://nanoworld88.narod.ru/pdb/20120208_001.pdb
Вести с ускорителей и не только!
lissagu: Добрый день, Александр!
У нас есть несколько источников на разную мощность и на разный диапазон. Типы TWT и MR-Klystron. Не могли бы Вы уточнить примерную мощность (+- 2-3 квт) и диапазон? И какой тип нужен: импульсный или непрерывный. Если импульсный - какова длительность импульса и характеристики его фронтов?
Кушелев: Я не знаю, какая минимальная мощность позволит включить рубиновый генератор, поэтому лучше начать эксперимент с максимальной мощности, которая у Вас есть. Сферический резонатор в Дубне светился. Мощность пучка 100 МВт, но в резонатор попадало меньше мегаватта однозначно. Может быть хватит и 1 киловатта, но заранее сказать затрудняюсь.
Диапазон желательно 5...8 мм. Это - наиболее удобный диапазон для изготовления резонаторов в моих условиях. Очень желательна плавная перестройка частоты хотя бы на уровне +-0.5ГГц. Когерентность должна быть не хуже ухода фазы на 90 градусов за длительность импульса. Длительность импульса >40 микросекунд.
Это - желаемые параметры. В реальности обычно всё хуже...
Я могу изготовить девайсы под Ваши параметры.
Ведём переговоры с потенциальными партнерами
abcd: ... я бы хотел ознакомиться с теоретической базой, выкладками, или прикидочными расчётами данного источника энергии. Не обязательно приводить точный расчёт, достаточно простых абстрактных формул типа E=(mV^2)/2, то-есть без учёта внешних факторов. Как показала практика, в некоторых случаях необходимо подбирать эти коэффециенты на основании экспериментов. Этого вполне хватит чтобы оценить соответствие возможностей оборудования требуемым параметрам и, при положительном соответствии, взять на этой основе финансирование. К сожалению, бюджет не очень большой, так как это частная инициатива, но старт-ап в районе 120 тыс. евро в месяц вполне реален ...
Kushelev: Предлагаю Вам следующий вариант взаимодействия ...
С уважением, Ваш А.Кушелев
Приглашение к сотрудничеству
Лаборатория Наномир готова к любому взаимовыгодному сотрудничеству. У нас есть сторонники как явные, которые помогают морально и материально, есть очень много пассивных наблюдателей, есть и ярые противники, которые используют любые методы и средства (аморальные и просто преступные), чтобы уничтожить работу лаборатории и дискредитировать ее.
В одиночку внедрить технологии, выводящие цивилизацию на новый уровень, невозможно. Благодаря поддержке множества заинтересованных людей проделана огромная работа. Ознакомиться с её результатами можно изучив материал рассылки "Новости лаборатории Наномир". Люди науки могут
изучить научные труды.
Вклад каждого не останется незамеченным в случае успеха в реализации научных проектов. Результаты совместной деятельности принадлежат участникам проекта пропорционально коэффициентам творческого и финансового участия.
Основные разработки:
Микроволновый источник энергии
В 2011 году были куплены рубиновые шарики для эксперимента на сумму ~1000 долл. В результате было сделано научное открытие, проверена защита диэлектрических резонаторов от перенапряжения. В этом году, вероятно, можно будет создать микроволновую энергетику, т.к. удалось найти сырьё (рубин #8), из которого сделаны рубиновые шарики для эксперимента в Дубне.
Параллельно началась серия экспериментов на ускорителе "B" в диапазоне 20 ... 140 ГГц.
"Эликсир вечной молодости"
28 сентября 2011года в институте геронтологии (г.Киев) начался эксперимент по созданию "эликсира вечной молодости". Благодаря первому взносу (в размере 500 долларов) Золдракса и поддержке других соинвесторов. Продолжаются переговоры с потенциальными инвесторами по поводу финансирования этого проекта.
Программа исследований Презентация
Пикотехнология
Созданы первые версии пикотехнологии (выпуски рассылки 212 270 271 272 ), с помощью которой Александр Кушелев и Виктория Соколик сделали более10 научных открытий.
Виктория Соколик: Уважаемые коллеги, Вашему вниманию предоставляется услуга -- моделирование 2D и 3D структуры любого белка без ограничений в его размере и степени изученности с помощью программного обеспечения, базирующемся на принципиально новом подходе декодирования нуклеотидной последовательности, детерминирующей данный белок.
Всё, что необходимо от заказчика, это нуклеотидная последовательность мРНК интересующего его белка (или код этой нуклеотидной последовательности в EMBL, или хотя бы код самого белка в PDB).
В течение 1-3 суток мы готовы предоставить Вам схему вторичной структуры заказанного белка (2D), модель его пространственной структуры (3D) в виртуальном пространстве, а также файл .pdb с координатами каждого атома белка.
Файл .pdb может быть использован по аналогии с файлами закристаллизованных белков из PDB банка для дальнейшего конформационного анализа белка методами молекулярной динамики с учётом физико-химической специфики микроокружения белка или его взаимодействия с лигандами.
Таким образом, Вы сможете максимально быстро удобным для Вас способом (по электронной почте, на сайте либо на электронном носителе) получить информацию о структурном шаблоне Вашего белка.
Первые 10 заказов -- бесплатно . Цена следующих заказов -- договорная.
Сотрудничество может быть различным:
- участие в научных дискуссиях на форуме (конструктивное)
- совместное создание коммерческого продукта
- поиск инвесторов
- выступить менеджером по продаже готовых коммерческих продуктов
- конструктивные предложения по продвижению идей лаборатории Наномир
- содействие в проведении экспериментов
и т.п.
- написание совместных научных статей и т.п.
- материальный вклад (денежный или обеспечение оборудованием и материалами)
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир.
+7-926-5101703, +7-903-2003424, +7-916-8265031, Skype: Kushelev2009, mail: kushelev2011@yandex.ru
веб-мани: WM-кошелек R426964799301