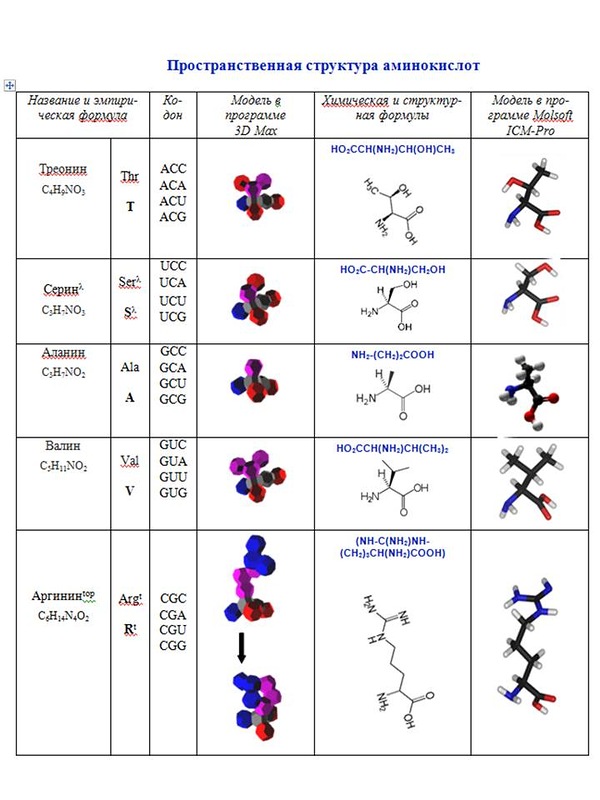
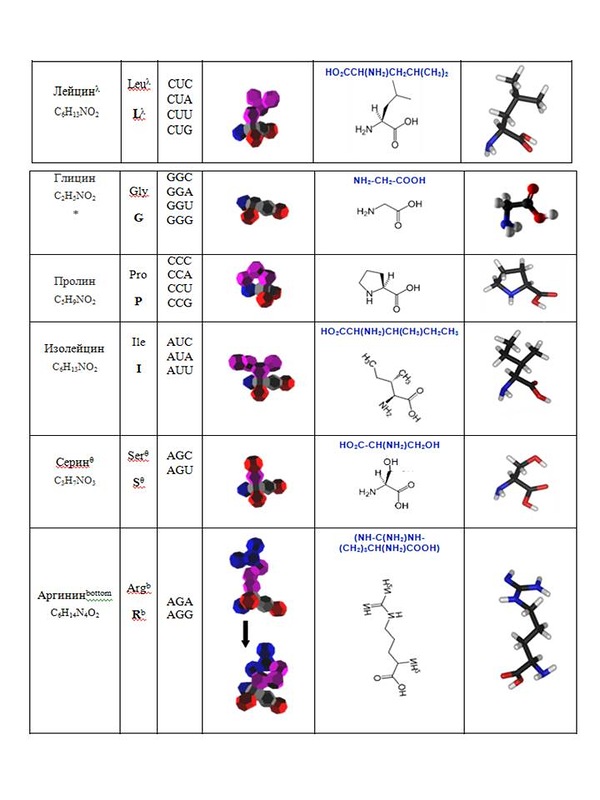
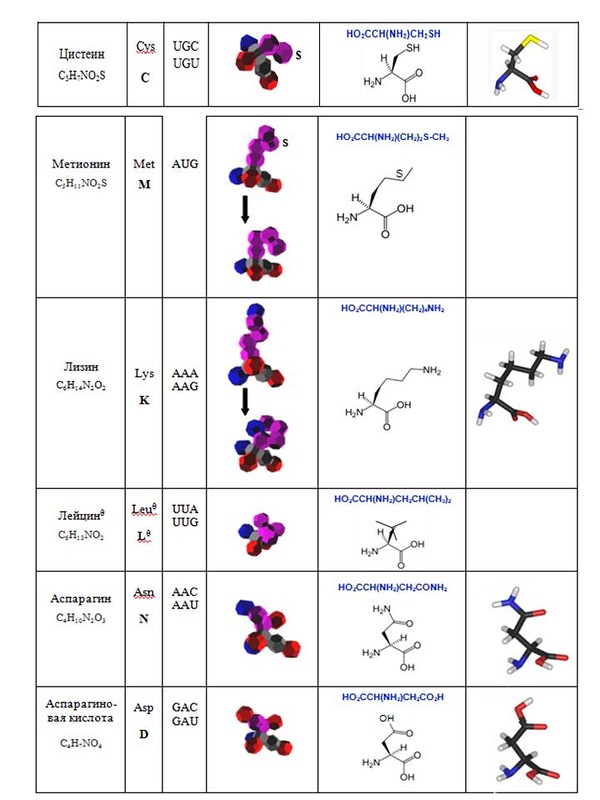
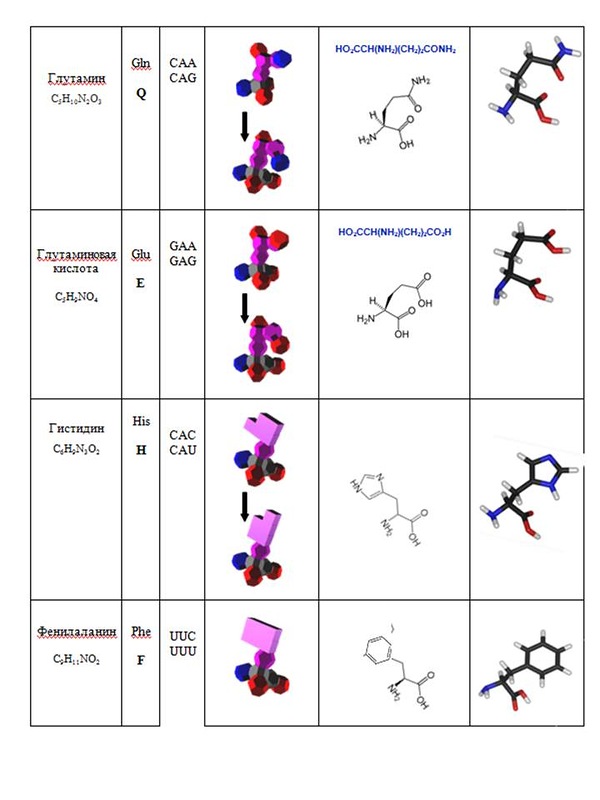
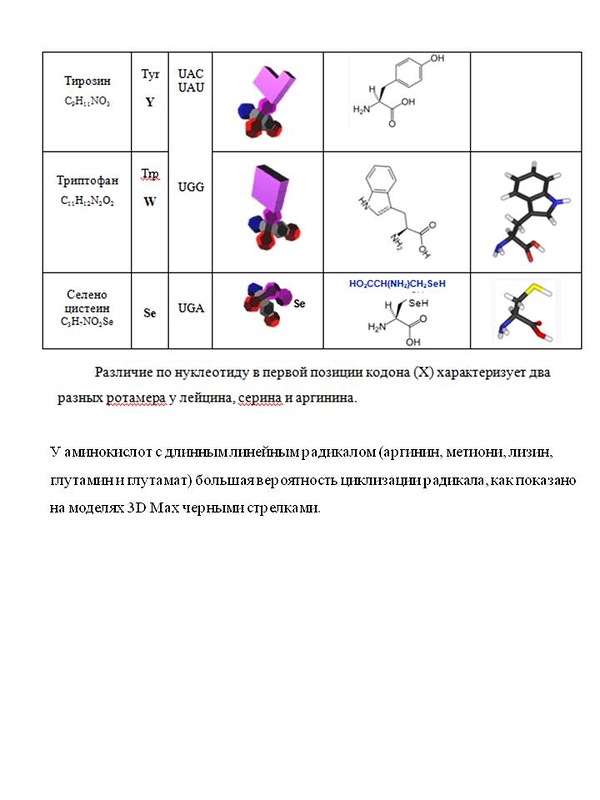
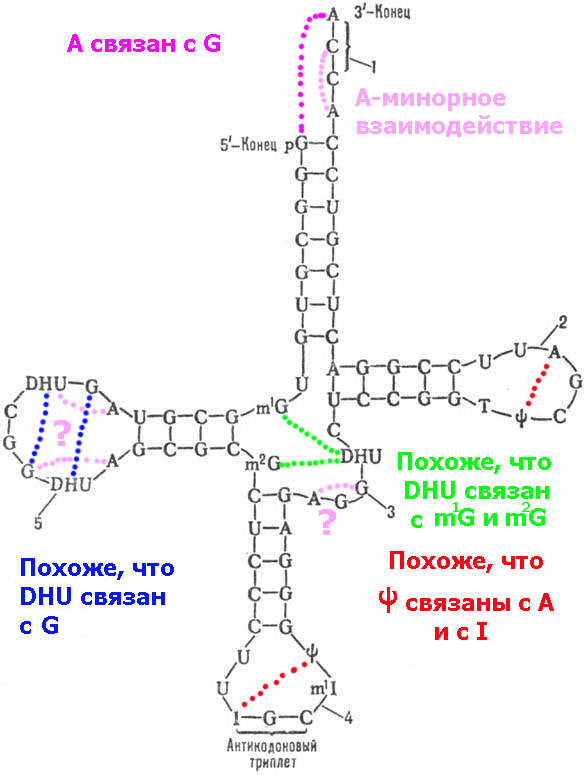
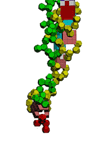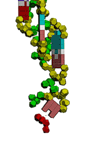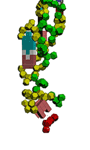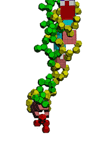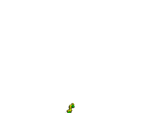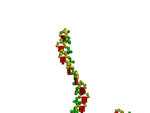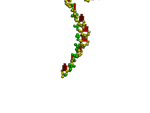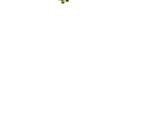Кушелев: О! Класс! Нужно будет выпустить рассылку на эту тему...
Но мы с
Вами помнится обсуждали ориентацию аминокислоты на акцепторном конце
тРНК. И эта ориентация, как мы выяснили, вдоль оси вращения тРНК. Вы
вроде согласились...
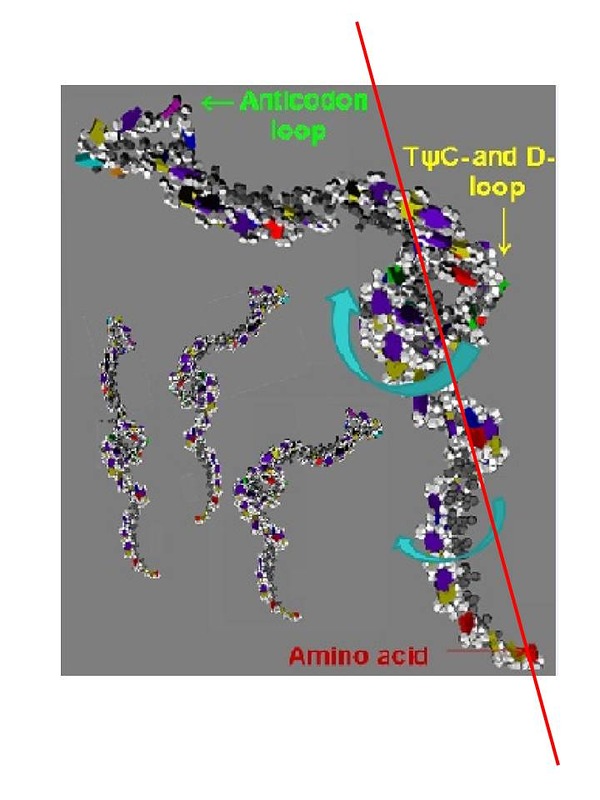
Но это возможно лишь в том случае, если молекула тРНК симметрична относительно оси симметрии акцепторного стебля:
Уважаемая
Виктория! Определитесь, пожалуйста, как в Вашей модели ориентирована
аминокислота и вокруг какой оси происходит вращение аминокислоты и
вместе с ней тРНК. Ведь от этих "мелочей" зависит, правильно ли Вы
представляете себе структуру тРНК или нет...
Класс! И всё же хотелось бы разобраться с выбором изомеров. Помнится,
Вы согласились, что альфа-спираль белка правая. В этом случае
получается такая модель из глицинов:
Если
же Вы замените модели аминокислот на другие (свой вариант), то у Вас
водородные связи не дотянутся, как мы уже разбирались ранее... Так
что желательно до передачи моделей программисту уточнить форму моделей
аминокислот. Иначе потом может потребоваться переделка...
Кушелев: Прекрасно! Значит ось вращения модели аминокислоты у Вас совпадает с
осью симметрии акцтепторного стебля. Это уже очень хорошо. Теперь
давайте разберёмся с механизмом трансляции. Помните, как мы проверяли
гипотезу, хватит ли кинетической энергии тРНК, чтобы срезать триплет
иРНК? Помните, что энергии хватило? А это значит, что гипотеза
подтвердилась. Следовательно, тРНК имеет симметричную форму и ось
симметрии тРНК одновременно является осью симметрии акцепторного (и
антикодонового) стебля.
Кушелев: Ну что же, давайте посмотрим на данные РСА, откуда следует,
что ось симметрии акцепторного стебля не проходит вдоль оси
антикодонового. Кстати, такие данные могут говорить лишь о существовании
L-образной конформации тРНК в кристалле. А при кристаллизации тРНК
могут "склеивать ласты", поэтому о конформации тРНК в растворе РСА
прямых данных дать не может принципиально. Согласны?
Поэтому
приходится косвенными способами выяснять, какой формы тРНК существует в
растворе. Гипотетический механизм трансляции требует, чтобы тРНК в
растворе имела крестообразную форму:
В
этом случае понятно, как работает механизм трансляции, начиная от
транспортировки аминокислот и заканчивая механикой композиционного
кодирования, т.е. управления углом поворота тРНК (и аминокислоты) с
помощью генетического кода.
В Вашем варианте механизм трансляции остаётся за кадром, что не есть хорошо. Согласны?
Кушелев: Мы же с Вами разбирались с альфа-спиралью белка. Из Ваших моделей
аминокислотных остатков она не получается. Водородные связи между
группами азота и кислорода не дотягиваются... И группы кислорода в Вашей
модели находятся снаружи альфа-спирали, а экспериментально установлено,
что они внутри, т.е. ближе к оси симметрии, чем группы азота.
Victoria пишет: мои представления в основном не противоречат экспериментальным данным.
Кушелев: "В основном" не считается. Любое противоречие может
опровергнуть гипотезу. Так что "в основном не противоречит" звучит
приблизительно как "в среднем по больнице температура нормальная" 
Victoria пишет:я же
уверена, что специфичность формы изоакцепторных тРНК обусловливают
синтез белка в рибосоме не только в виде правой альфа-спирали, но и в
виде бета-тяжа, витка левой 3-10 спирали или неупорядоченного фрагмента
без элементов вторичной структуры,
Кушелев: Тут тоже скрывается противоречие. Если композиционный код
существует, т.е. существует механизм композиционного кодирования, то он
работает для всех кодов. Т.е. нельзя половину пути ехать с рулевым
управлением, а половину - без руля. Согласны? Это значит, что все
фрагменты белка упорядочены. Другое дело, что порядок в виде
альфа-спирали иногда заметен на уровне РСА, а, скажем, участок спирали
коллагена РСА не обнаруживает, т.к. коллаген не кристаллизуется, как,
впрочем и фиброин. Но это не значит, что у коллагена и фиброина нет
упорядоченной структуры. Просто РСА не может её обнаружить. Но другие
методы позволяют. Это же относится и к тем фрагментам, которые Вы
считаете неупорядоченными. Порядок есть, просто он может быть непонятен 
Вы
видели материал по перевёртышам? Вы согласны, что перевёртыши не могли
бы существовать, если бы их структура не являлась упорядоченной?
Подробнее
Перевёртыши подтверждают, что вся структура белка без исключения является
упорядоченной. Согласны?
Victoria пишет:сама тРНК при трансляции НЕ ВРАЩАЕТСЯ. Угол поворота УЖЕ реализован в её структуре и помечен первым нуклеотидом антикодона.
Кушелев: И это мы с Вами обсуждали. При движении тРНК вращается. Она будет
вращаться даже в том случае, если "склеит ласты" и станет L-образной.

Чтобы
в этом убедиться, достаточно посмотреть на бумеранг той же L-образной
формы. Или семена клёна. При падении они начинают вращаться.
Когда
становится ясно, что тРНК при движении вращается, возникает гипотеза:
"тРНК вращается вокруг оси симметрии, совпадающей со связью C-N
аминокислоты. Именно вращение вокруг этой оси и позволяет реализовать
композиционный генетический код.
А дальше выясняется, что в
процессе движения "крылья" тРНК могут подтормаживать в зависимости от
интенсивности сигнала рибосомы, т.е. чем ближе к рибосоме, тем сильнее.
Это позволяет направлять тРНК к рибосоме с помощью гиперакустического
сигнала.
Пикотехнологическая модель антикодоновой петли тРНК
показала, что первый нуклеотид антикодона (инозин) при стыковке с
триплетом иРНК точно попадает на диэфирную связь и может её перерезать,
если хватит кинетической энергии тРНК. Мы с Вами проверили. Кинетической
энергии тРНК хватает, чтобы разорвать диэфирную связь.
Пикотехнологическая модель показывает, что после разрыва фосфодиэфирной
связи триплет иРНК продолжает вращаться вместе с тРНК вокруг
той самой оси симметрии. При этом остановить вращение можно с помощью
комплементарного нуклеотида, входящего в состав рибосомальной РНК. Это
позволяет реализовать композиционный генетический код.
Если у Вас есть альтернатива, то буду рад ознакомиться и сравнить все "за" и "против".
А
пока мне неизвестна альтернатива механизма композиционного кодирования.
Я правильно понял, что Вы альтернативы не предлагаете? 
Victoria пишет:Очень даже получается
Ну так покажите. Интересно посмотреть, какая длина водородных связей в
Вашей модели альфа-спирали, и какие атомы находятся ближе к оси
симметрии, кислорода или азота?
(смотреть через зеркало)
В
моей модели мы видим нормальной длины водородную связь. Атомы кислорода
находятся ближе к оси симметрии альфа-спирали, а атомы азота дальше.
Victoria пишет:Все
противоречия заключаются в том, что Вы не хотите абстрагироваться от
придуманной Вами гипотезы вращения тРНК в виде волчка и очень
гипотетического зова рибосомы  .
.
Даже
если она и вращается каким-то образом в цитоплазме наряду с другими
формами броуновского движения (учтите очень большую вязкость
цитоплазмы), то в рибосоме ей просто негде повернуться. Там слишком
узкая щель между субъединицами рибосомы для контакта антикодона тРНК с
кодоном мРНК. Поэтому каждая (или любая) тРНК взаимодействует с
рибосомой ЕДИНООБРАЗНО с одними и теми же группами атомов, а поворот
акцепторного стебля с аминокислотой на конце УЖЕ реализован в структуре
тРНК. Для этого собственно и были изобретены эволюцией изоакцепторные
тРНК. Кстати, обратите внимание, что благодаря инозину в первой позиции
антикодона тРНК (вместо цитозина и гуанина) кодоны с цитозином и
гуанином в третьей позиции прочитываются одними и теми же тРНК. Это
значит, что транслируется одна и та же конфигурация пептидной связи (R) и
синтезируется одна и та же вторичная структура, а именно правая
альфа-спираль. В Вашем варианте они прочитываются по разному и
синтезируются различные правые спирали или одна смешанная спираль. Т.е.
код пространственной структуры белка всё-таки троичен (три разновидности
углов поворота и конфигураций пептидной связи), а не квартетен, как в
Вашем композиционном коде.
Victoria пишет:в
рибосоме ей просто негде повернуться. Там слишком узкая щель между
субъединицами рибосомы для контакта антикодона тРНК с кодоном мРНК.
Кушелев:
А давайте сравним размер щели с размером тРНК. Если Ваша гипотеза
подтвердится, т.е. щель окажется меньше, чем размер тРНК, то мне
придётся пересмотреть механизм композиционного кодирования.
Ну что, конкретные размеры есть? 
Victoria пишет:благодаря
инозину в первой позиции антикодона тРНК (вместо цитозина и гуанина)
кодоны с цитозином и гуанином в третьей позиции прочитываются одними и
теми же тРНК.
Кушелев:
Вы согласны, что первый нуклеотид антикодона (инозин) точно попадает
на фосфодиэфирную связь и ... отрезает её при стыковке антикодона тРНК с
кодоном иРНК? Если нет, то давайте посмотрим на Вашу, т.е.
альтернативную модель антикодоновой петли тРНК.
Если
альтернативной пикотехнологической модели антикодоновой петли у Вас нет,
то придётся смириться с моим вариантом механизма композиционного
кодирования 
Victoria пишет:...
благодаря инозину в первой позиции антикодона тРНК (вместо цитозина и
гуанина) кодоны с цитозином и гуанином в третьей позиции прочитываются
одними и теми же тРНК. Это значит, что транслируется одна и та же
конфигурация пептидной связи (R) и синтезируется одна и та же вторичная
структура, а именно правая альфа-спираль. В Вашем варианте они
прочитываются по разному и синтезируются различные правые спирали или
одна смешанная спираль. Т.е. код пространственной структуры белка
всё-таки троичен (три разновидности углов поворота и конфигураций
пептидной связи), а не квартетен, как в Вашем композиционном коде.
Кушелев: В моём механизме композиционного кодирования тРНК может
повернуться на любой угол. Остановить вращение может комплементарный
третьему нуклеотиду триплета иРНК нуклеотид, входящий в структуру
рибосомальной РНК. Давайте посмотрим, сколько таких нуклеотидов
расположено в рибосоме вдоль траектории движения третьего нуклеотида
триплета иРНК. Если их окажется три, то Вы правы. Если их окажется
четыре, то прав я.
За структуру рибосомы в 2009-ом году дали
Нобелевскую. Давайте посмотрим, сколько в этой структуре нуклеотидов по
круговой траектории расположено. 3 или 4? Если 3, Ваша взяла, если 4 -
моя 
Victoria пишет:не
ИНОЗИН в первой позиции антикодона тРНК, а МЕТИЛИНОЗИН по-соседству с
антикодоновым триплетом гипотетически может разрезать фосфодиэфирную
связь в мРНК.
Кушелев: Совершенно верно. Не инозин, а метилинозин. Спасибо за поправочку 
Victoria пишет:А.Ю.,
Вы запутались. Посмотрите на свои первые посты в этой теме, в которых Вы
пишите, что не ИНОЗИН в первой позиции антикодона тРНК, а МЕТИЛИНОЗИН
по-соседству с антикодоновым триплетом гипотетически может разрезать
фосфодиэфирную связь в мРНК.
А теперь внимание: только у единственной
изоакцепторной тРНК аланина в этой позиции метилинозин (см. базу
первичных структур тРНК: http://trnadb.bioinf.uni-leipzig.de/Sea … vTax=2759).
Поэтому обобщать данное исключение до общего правила НЕ ВЕРНО.
Попробуйте
смоделировать Вашу модель разрезания для остальных нуклеотидов, которые
встречаются по-соседству с триплетом антикодона, и Вы убедитесь, что
заблуждались.
Кушелев: Очень любопытно. Давайте посмотрим схемы антикодоновых петель других тРНК
Действительно,
вместо метилинозина в этом месте может быть, например, метилгуанин. Но
он так же может резать диэферную связь, как и метилинозин.
Интересно, а как в эксперименте различают, где находится инозин, а где - метилинозин? Могут их перепутать?
***
Кушелев: В моём механизме композиционного кодирования тРНК может
повернуться на любой угол. Остановить вращение может комплементарный
третьему нуклеотиду триплета иРНК нуклеотид, входящий в структуру
рибосомальной РНК. Давайте посмотрим, сколько таких нуклеотидов
расположено в рибосоме вдоль траектории движения третьего нуклеотида
триплета иРНК. Если их окажется три, то Вы правы. Если их окажется
четыре, то прав я.
За структуру рибосомы в 2009-ом году дали
Нобелевскую. Давайте посмотрим, сколько в этой структуре нуклеотидов по
круговой траектории расположено. 3 или 4? Если 3, Ваша взяла, если 4 -
моя 
Виктория: С удовольствием посмотрю как Вы их будете искать
Кушелев: Это непросто, т.к. точность определения структуры рибосомы "оставляет
желать лучшего", т.е. погрешность модели 2009 года достигает 50
нанометров, а для проверки нашей гипотезы нужна пикотехнологическая
точность, т.е. хотя бы 100 пикометров, что на два порядка точнее...
***
Victoria пишет:
Рекомендую ознакомиться с любопытной трактовкой эволюции пространственной структуры тРНК
Кушелев: Очень интересная статья!
Цитата: ... Майцелс и Вайнер
предположили, чтот-РНК-подобная структура древних РНКовых организмов
состояла из коаксиальных T-петли и акцепторного стебля [Maizels N.,
Weiner A.M. 1994]. Это предположение основано на двух группах известных
фактов.
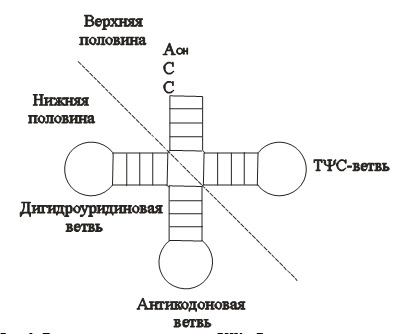
Во-первых, верхняя половинасовременных т-РНК является
независимым структурным доменом, который узнается РНКазой P (один из
ключевых молекулярных реликтов, предположительно существовавших ещё в
мире РНК, о ней подробнее далее в статье), фактором элонгации Tu,
АРСазами и даже рибосомными РНК. Важность этой части т-РНК почти во всех
макромолекулярных взаимодействиях дает основание считать, что она более
древняя, чем нижняя половины молекулы.
Во-вторых, способность
клеток различать т-РНК разных видов зависит, преимущественно
отспецифических нуклеотидов верхней половины молекулы т-РНК, включая
«дискриминаторное основание» (это комплементарная пара оснований,
находящаяся в полжении 2-72 (о нумерации см. Аминоацил-тРНК-синтетазы -
два класса ферментов) и являющаяся ключевой для распознавания АРСазами).
Авторы оставляют открытым вопрос о возникновении нижней половины тРНК
(рис.1).
Конец цитаты.
Кушелев: Статья очень ценная. Похоже, что авторы докопались до нюансов эволюции тРНК...
Действительно похоже на то, что древние тРНК фактически выполняли
функцию рибосомы, т.е. каждый тип нёс свою аминокислоту и присоединял в
растущую белковую цепь у ДНК, а позднее у иРНК. Но, постепенно система
белкового синтеза усложнялась...
Кушелев: Действительно,
вместо метилинозина в этом месте может быть, например, метилгуанин. Но
он так же может резать диэферную связь, как и метилинозин.
Виктория: Метилгуанин в данной позиции такой же раритет, как и метилинозин.
Вы
что даже без моделирования, на авось, утверждаете, что любой нуклеотид
будет работать как метилинозин и даже маленький цитозин? Проверьте
пожалуйста для начала Вашу гипотезу для всех встречающихся в данном
положении нуклеотидов.
Кушелев: Я бы сначала хотел разобраться, как выясняют, что тот или иной нуклеотид
находится в первой позиции антикодона. Пикотехнологическая модель
показывает, что этот нуклеотид находится в месте диэфирной связи.
Естественно, что этот механизм может работать только на уровне эукариот,
хотя может быть работает уже и на уровне прокариот. Но не на уровне
митохондрий с их более древним генетическим кодом.
Глядя на ряд схем тРНК нетрудно догадаться, что точность расположения
нуклеотидов в них оставляет желать лучшего. Ведь на схеме акцепторный
(АСС) конец разомкнут. Пикотехнологическая модель показывает, что он
замкнут. При этом положение крайнего по схеме аденина отличается от
положения в пикотехнологической модели на десятки нанометров. Это
говорит о том, что положение нуклеотидов в антикодоновой петле тРНК на
схеме тоже обозначено приблизительно, т.е. с точностью до нескольких
десятков
Пикотехнологическая
модель акцепторного конца тРНК интересна тем, что в ней наблюдается ряд
совпадений, которые убеждают, что геометрия этого участка тРНК именно
такая. При другой геометрии группа азота просто не попадёт на ось
вращения, совпадающую с осью симметрии акцепторного стебля. Кстати,
интересно было бы увидеть Ваш вариант акцепторного стебля с
аминокислотой на конце. Попадает ли у Вас группа азота на ось симметрии
акцепторного стебля?
На Вашей схеме ось симметрии видна, а аминокислоты с группой азота что-то не видно...
Вы не могли бы показать эту зону тРНК крупнее?
Victoria пишет:Посмотрите пожалуйста пост 83 и 86. Вы же ещё анимацию делали из моих фото и уже выясняли этот вопрос.
Кушелев: На
пластмассовой модели плохо видна ориентация аминокислоты и расстояние
от центра атома азота до оси симметрии акцепторного стебля. Я предлагаю
сделать компьютерную модель, точнее увеличить масштаб изображения Вашей
модели:
и закрутить её вокруг этой оси в программе 3DS Max
Как-то так:
Мне
просто очень интересно выяснить, может ли центр атома азота снова точно
попасть на ось вращения? Если не попадёт, то выбор между моделями будет
очевиден 
Victoria пишет:А.Ю.,
давайте лучше вспомним скрипты для тРНК и попытаемся добиться того,
чтобы после поворота нуклеотидной цепи в любой петле цепи сходились в
двойную спираль.
Кушелев: Это дело тонкое. Об этой проблеме я писал в 230-ом выпуске рассылки "Новости лаборатории Наномир": http://nanoworld88.narod.ru/data/230.htm
Без
моделирования транспозиционных углов проблема не решается. А для их
моделирования нужно привлекать программистов. Я в программировании
дилетант, поэтому на меня мало надежды...
Кстати,
энергия пяти водородных связей скорее всего отличается от энергии
диэфирной связи. Это может помочь для проверки моей модели АСС-конца
тРНК. Давайте прикинем, на сколько должна отличаться энергия связи по
моей модели от стандартной, т.е. из современного учебника молекулярной
биологии.
Итак, в Википедии читаем: "Энергия водородной связи значительно
меньше энергии обычной ковалентной связи (не превышает 40 кДж/моль)"
Цитата: Водородная связь (4-120 кДж/моль) Конец цитаты.
Цитата:
фосфодиэфирная связь (относится к категории прочных ковалентных связей). … составляет около 30,6 кДж/моль. Конец цитаты.
Цитата: энергия ковалентной связи — 400—900 кДж/моль. Конец цитаты.
Обнаружилась некая нестыковочка в цифрах, но мы сейчас уточним...
Примем пока величину энергии фосфодиэфирной связи за 30,6 кДж/моль
Цитата: Энергия водородной связи Н---О в димере воды (H2O)2 составляет 21,5 кДж/моль Конец цитаты.
Вероятно,
энергия водородных связей, которыми удерживается аминокислота, не
превышают энергию водородной связи в димере воды. По максимуму
получается 21.5*5=107.5 кДж/моль, а по минимуму может быть в несколько
раз меньше, т.е. может быть даже 30 кДж/моль. Ведь минимальная энергия
водородной связи всего 2 кДж/моль...
Таким образом нужно проводить
дополнительное исследование, чтобы уточнить, какая энергия требуется
для отделения аминокислоты от АСС-конца тРНК. Если эта энергия известна,
то её легко сравнить с энергией фосфодиэфирной связи (30.6 кДж/моль)
Какова же энергия аминоацилирования?
Цитата: ... на присоединение аминокислоты к полипептидной цепи затрачивается около 90 кДж/моль Конец цитаты.
Судя по этой цитате:
"При аминоацилировании тРНК затрачивается энергия одной связи молекулы
АТФ", энергия аминоацилирования не измерялась в эксперименте.
Так что придётся измерить и сравнить с энергией фосфодиэфирной связи. Если не сойдётся, то старой теории хана...
Цитата: на синтез аминоацил-тРНК затрачиваются две богатые энергией фосфатные связи. Конец цитаты.
Кушелев: Вот оно и выясняется...
А вот и объяснение нестыковочки:
Цитата: Суммарная реакция превращений, приводящих к образованию аминоацил-тРНК, может быть записана следующим образом:
Аминокислота + АТР + тРНК + Н2О --> Аминофцил-тРНК + AMP + 2Pi
Из
этой реакции следует, что на синтез аминоацил-тРНК затрачиваются две
богатые энергией фосфатные связи. Одна из них расходуется на образование
эфирной связи между аминокислотой и тРНК, а другая (при гидролизе
пирофосфата) сдвигает равновесие всей реакции в сторону образования
продукта.
Конец цитаты.
Короче, баланс энергии не сходится вдвое...
А это значит, что реальная энергия связи аминокислоты с АСС-концом тРНК
приблизительно вдвое больше энергии фосфодиэфирной связи и составляет
около 60 кДж/моль.
При этом на каждую водородную связь приходится в среднем по 12 кДж/моль.
Victoria пишет:Для
образования водородных связей нет нужды затрачивать энергию АТФ.
Представьте себе какими энергозатратными процессами тогда стало бы
формирование двойных спиралей нуклеиновых кислот или вторичных структур
белков. На самом деле их формирование сопровождается выигрышем в энергии
не напряженных структур. Более того, реакции сопряженные с распадом АТФ
-- необратимы, а фолдинг или денатурация-ренатурация белка процессы
обратимые.
Поэтому аминокислота присоединена к АСС-стеблю тРНК
обычной одинарной ковалентной связью, а не пятью водородными как
придумали Вы. Более того, на её формирование затрачивается энергия
только одной макроэргической фосфодиэфирной связи АТФ (34 кДж/моль),
энергия второй (как Вы верно цитировали выше) нужна только для сдвига
обратимой реакции в сторону продукта и следовательно не аккумулируется в
образующейся связи между аминокислотой и рибозой тРНК.
Вы со мной согласны, Александр Юрьевич?
Кстати,
для сравнения: при гидролизе пептидной связи выделяется только 2
кДж/моль, а распаде глюкозы -- 17,6 кДж/моль. Время жизни любой молекулы
АТФ не более 1 минуты.
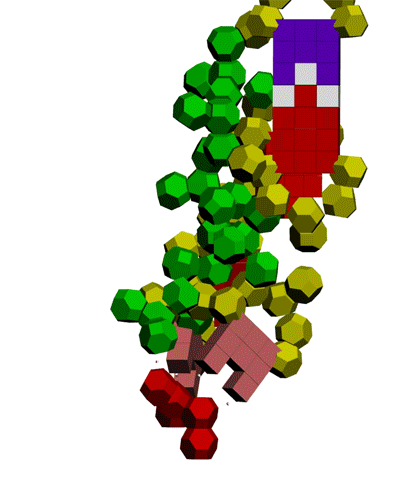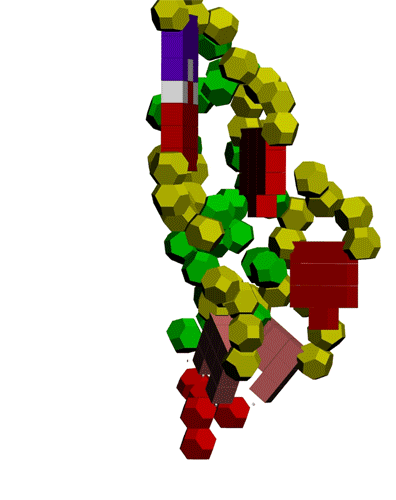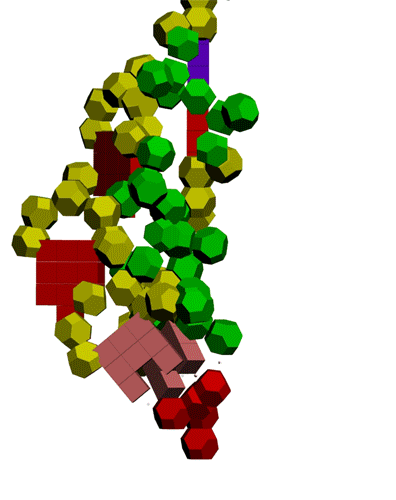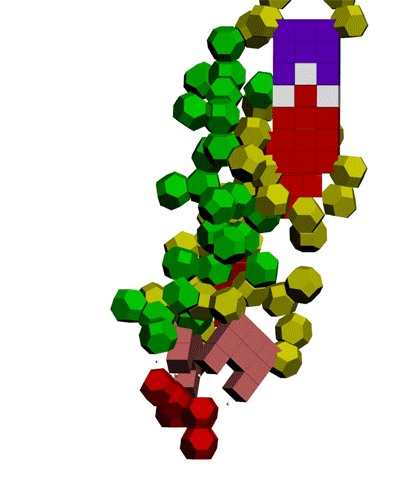
Кушелев: Давайте
ещё раз посмотрим на пикотехнологическую модель АСС-конца тРНК с
присоединённой аминокислотой. Очевидно, что присоединение и отсоединение
аминокислоты требуют затрат энергии, хотя энергетический баланс
"присоединить плюс отсоединить" равен нулю. Другое дело, что затраты
зависят от энергии связи. Чем она больше, тем больше и затраты.
Как
я понимаю, отсоединение аминокислоты, согласно стандартной модели,
сопровождается не поглощением, а выделением энергии. Той самой энергии,
которая запаслась при образовании фосфодиэфирной связи.
Согласно
моей модели присоединение аминокислоты напротив сопровождается
выделением энергии. А при отсоединении тРНК от достроенной белковой цепи
требует затрат энергии от рибосомы.
Почему же тогда поглощается ~60 кДж/моль энергии при амилировании тРНК?
Давайте разберёмся. Откуда взялась эта цифра? Может быть это вообще гипотеза? 
В
реальности процесс амилирования тРНК может сопровождаться выделением
энергии в результате образования 5 водородных связей. Но это - мелочь,
по сравнению с энергией, которая запасается при движении тРНК за счёт
хаотического движения молекул физраствора. Мы уже разобрались, что
кинетическая энергия тРНК как минимум на порядок превосходит энергию
ковалентной связи. Эта энергия используется для отрезания триплета иРНК и
реализации композиционного генетического кода. В процессе наращивания
белковой цепи на один аминокислотный остаток выделяется энергия, которая
в т.ч. может разрывать водородные связи, удерживающие аминокислотный
остаток АСС-концом тРНК. Таким образом картина амилирования тРНК и
синтеза белка может выглядеть иначе и более логично, чем в современном
учебнике молекулярной биологии. Согласны?
А от Вас хотелось бы
увидеть укрупнённо АСС-конец тРНК с аминокислотой. Удастся ли Вам, глядя
на современный учебник молбиологии разместить атом азота на оси
симметрии акцепторного стебля тРНК?
В моей пикотехнологической
модели это произошло автоматически. Интересно посмотреть, что получится у
Вас. Если не получится, то придётся пользовать мою модель АСС-конца (и
тРНК вцелом). Согласны?
Victoria пишет:Энергия, которая необходима для формирования той или иной
химической связи не всегда совпадает со значением свободной энергии,
которая выделяется при её разрыве. Это детерминируется силой связи.
Поэтому Ваши расчеты нуждаются в уточнении, не говоря уже о самой модели
амилирования тРНК.
Я даже больше скажу, что образование одних связей требует затрат
энергии, а другие образуются сами с выделением энергии. Амилирование,
согласно моей модели, происходит с выделением энергии, а согласно
стандартной гипотезе требует затрат двойной энергии фосфодиэфирной связи

Интересно было бы проверить экспериментально, какая из двух моделей верна.
Victoria пишет:Укрупнённо АСС-конец тРНК с аминокислотой я смоделирую на недельке. Сейчас конец года, три отчета "горят" ещё на вчера  .
.
Кушелев: Буду ждать.
Для выбора модели это имеет большое значение. Чтобы
программисту потом не пришлось переделывать одну модель на другую.
***
Кушелев: Уважаемая Виктория!
А Вы не хотели бы в своих научных
статьях использовать модели, созданные мною в 3DS Max? В частности,
показать альтернативную Вашей пикотехнологическую модель тРНК?
Может быть это вызовет дополнительный интерес читателей к Вашим работам...
Victoria: я не могу обсуждать и тем более публиковать модели тРНК, которые считаю ошибочными.
Кушелев:
Почему, собственно? Ведь в процессе обсуждения находятся аргументы "за"
и "против", с помощью которых можно выбрать из нескольких моделей
наиболее адекватную. Нельзя же усилием воли выбрать правильную модель.
:)
Сначала Вы опубликовали неправильные модели азотистых оснований:

Потом Вы опубликовали правильные модели (вообще-то тоже не факт!).

Что в этом плохого?
Обсудить на форуме
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир.
+7-926-5101703, +7-903-2003424, +7-916-8265031, Skype: Kushelev2009, mail: kushelev2011@yandex.ru