|
Выпуск 271
Лаборатория Наномир
Когда реальность открывает тайны,
уходят в тень и
меркнут чудеса ... Аукцион высоких технологий лаборатории Наномир 
Существуют фирмы, которые выпускают конструкторы для изучения строения вещества. Очень красивые,
но ... устаревшие  Среди моделей есть и белки и тРНК ... Есть модели, объясняющие фолдинг (укладку) белка (по теме CASP!) Есть модели сложных белков типа антител, гистонов и шаперонов ... Есть даже модель рибосомы! Эти модели делают разные фирмы по разным технологиям. Что-то удобно делать штамповкой, что-то с помощью 3D-печати. Для этих (и не только!) фирм я хочу предложить аукцион научных разработок, в частности, пикотехнологических моделей элементарных частиц, электронных оболочек атомов и молекул, структур кристаллов, белков, ДНК/РНК, комплексов... Есть и фемтотехнологические модели элементарных частиц, мезонов, нуклонов, атомных ядер, экзотических
молекул типа Ps2, экзотических кристаллов типа мюонного и позитрониевого... Теоретически можно было бы самим заключить контракты с фирмами, которые распечатывают модели на 3D-принтерах, но у фирм, торгующих моделями, есть большая клиентская база, оборот. Поэтому целесообразно предложить новые, более точные модели в виде файлов непосредственно фирмам-изготовителям моделей и конструкторов. Они фактически являются заказчиками образцов наукоёмкой продукции. Учитывая, что таких фирм много, имеет смысл организовать для них аукцион пикотехнологических (и не
только) компьютерных моделей. Ассортимент моделей может быстро расти, но начать можно с тех же базовых моделей, что уже выпускаются. Просто перейти с нано-точности на пико-точность. Спрос на пико-точность непременно обгонит спрос на наноточность, ведь нано-точность, в действительности, не ангстремы, как считает большинство исследователей допикотехнологического мира. Нано-точность бывает хуже 50 нанометров. А точность в доли ангстрема - это рекорд для присталлов, причём не для всех  И это не значит, что с такой точностью можно определить начало или конец альфа-спирали. С такой точностью
можно определитьтолько середину. При этом нет гарантии, что в этой середине нет разрыва этой спирали И это не значит, что с такой точностью можно определить начало или конец альфа-спирали. С такой точностью
можно определитьтолько середину. При этом нет гарантии, что в этой середине нет разрыва этой спирали  Что же прелагается вместо старых, нанотехнологических моделей? Перед Вами новый, пикотехнологический модельный ряд: Простые пикотехнологические конструкторы, из которых можно собирать модели электронных оболочек и структуры атомов и простых молекул, уже продаются. Продаются и учебные пособия для них. А на аукцион можно предложить и модели посложнее. Например, гистоны
и шапероны. ДНК и РНК Фирмы, участвующие в аукционе, могут сами определиться, какие пикотехнологические модели атомов, молекул и кристаллов, углеводов, белков и ДНК, фемтотехнологические модели элементарных частиц, нуклонов, ядер... они хотели бы приобрести в первую очередь. Предлагаю активным участникам форума лаборатории Наномир найти наших
потенциальных заказчиков и собрать на аукционе высоких технологий лаборатории Наномир. Одну фирму-производителя таких моделей уже нашли на квант-форуме:http://www.3dmoleculardesigns.com/ Руководству этой и аналогичных фирм нужно разослать приглашения на аукцион. Обсуждение
на форуме лаборатории Наномир.
Сегодняшний предел электронного микроскопа  Комплекс прионных белков под трансмиссионным электронным микроскопом. (фото Eye of Science). Подробнее. Обсуждение.
Фаногория в Тамани 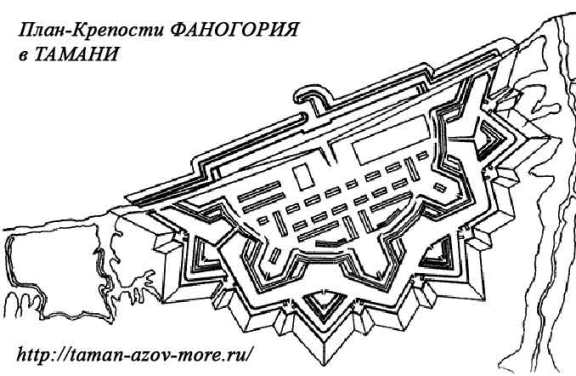 А этот мегалитический объект я пытался в своё время найти. 
Материал с форума лаборатории Наномир:Кушелев: Но кроме этого рисунка не смог найти изображений объекта. Svetlana555: Через гугл отлично видны остатки крепости даже сейчас. Кушелев: А координаты не дадите? Svetlana555: 45.22171,36.747208 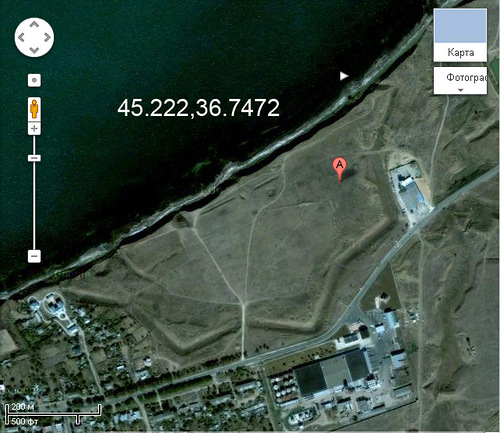 Координаты:
45.222,36.7472 Кушелев:
Ещё раз благодарю! Оказывается, неплохо сохранился... 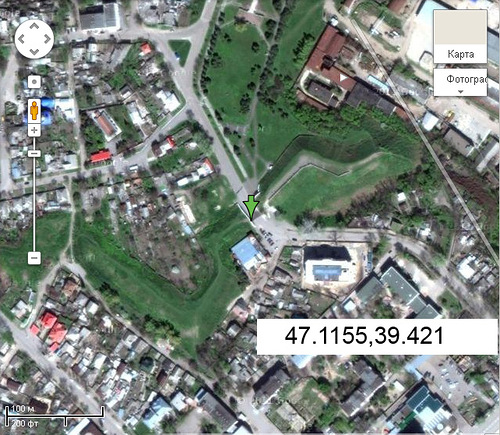
Координаты ещё одного выпаривательного комплекса: 47.1155,39.421 Интересно, каким был изначально этот мегалитический комплекс? Обсуждение.
Дискуссия о форме аминокислот Victoria: В посте № 558 этой темы был представлен предварительный анализ формирования такого элемента вторичной и третичной структур белка, как поворот. Дальнейшее моделирование на физической модели полипептида позволило предсказать формирование водородной связи, фиксирующей повороты 1-го и 2-го типов, и обобщить закономерности кодирования этих поворотов. В таблице на фото моделей поворотов 1-го и 2-го типа водородная связь обозначена голубой пунктирной линией. 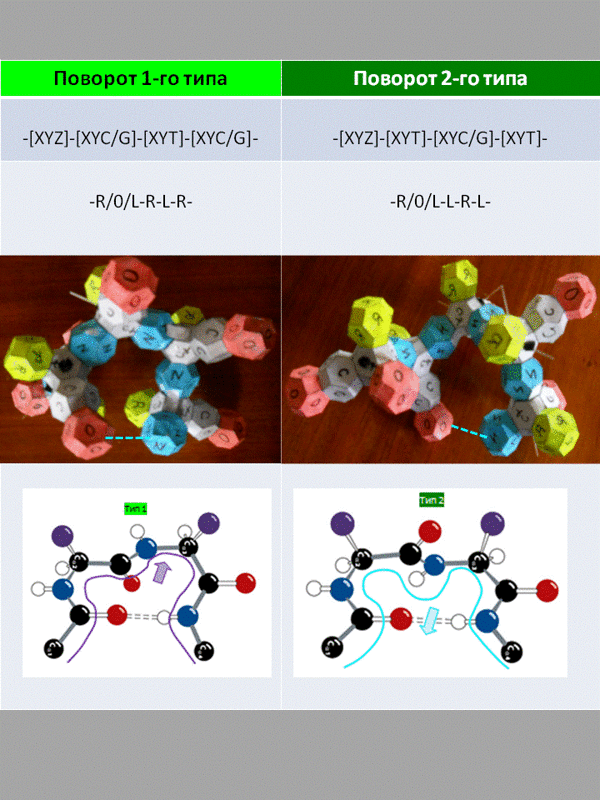
Кушелев: К сожалению, в Вашей модели группы =CO разных аминокислотных остатков направлены по-разному относительно оси симметрии альфа-спирали. Нужно исправить это недоразумение... Обсуждение.
Настраиваем
полновесный скрипт Савина-Кушелева Денис Савин: Setup Angles Dialog Это диалоговое окно позволяет настраивать систему углов в интерактивном режиме.Также удобно настраивать деформации для всей цепочки. Interface 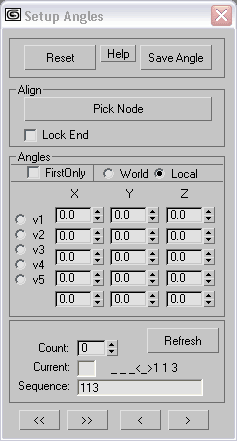
First group
Reset - Восстанавливает значения углов по умолчанию, из конфигурационого файла. Help - Открывает файл помощи. Save Angles - Сохраняет углы в конфигурационном файле для последуещего ипользования при построении. Align group
Pik Node - Позволяет указать обьект, по которому будет выравнена вся цепочка. Locк End - Указывает, что выравнивание будет производится по последнему остатку в цепочке, иначе выравнивание производится по первому остатку. Angles group
First Only -
Указывает, что при настройке угла, отображаться эти изменения будут только на последнем остатке, а все остальные останутся неподвижны.Чтобы увидеть все изменения внесенные этой настройкой, нужно обновить всю цепочку с помощью кнопки Refresh. World - Система координат, используемая программой при построении. Унаследованная от алгоритма A. Неделько. Далее не ипользуется из-за неудобства при настройке. Local - Система координат используемая для настройки углов.
В локальной системе координат нулевые углы для всех трех осей устанавливают два соседних остатка в начальное положение, от которого удобно начать настройку. При этом кольца в месте соединения расположены в одной плоскости, а ось Z перпендикулярна им, что позволяет легко
вращать один остаток относительно другого для выбора нужного угла. Оси X,Y при этом отвечают за наклон в ту, или иную сторону. 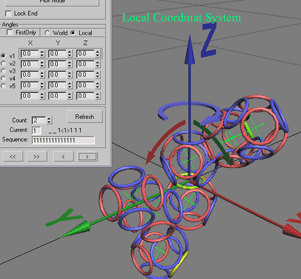
V1-5 - Варианты углов для соединения остатков в цепочку. Sequence group
Refres - Обновляет и перерисовывает всю цепочку. Count - Номер текущего остатка. Current - Вариант угла текущего остатка. Seqence - Последовательность
вариантов углов (1-5) по которым будет построенна цепочка. Если этот диалог вызывается из диалога Create Rings Atoms то последовательность углов будет взята у текущего на тот момент файла. Buttons
<< - Удалить все созданные остатки. >> - Создать все остатки из цепочки. < - Удалить последний созданный остаток. > - Создать следующий остаток. *** Create Rings Atoms Dialog Это диалоговое окно позволяет управлять всеми основными свойствами
построения кольцегранных моделей.Все настройки, произведенные в этом окне, будут атоматичиски сохранены и в дальнейшем, будут применяться по умолчанию. Interface 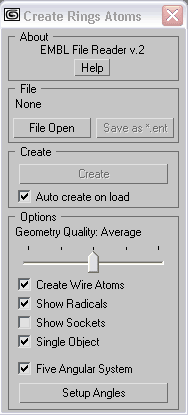
File group
File Open - Открывает файл совместимый со следующими базами данных: DDBJ/EMBL/GenBank. Если включена опция Auto create on load, то сразу после открытия начинается создание, иначе чтоб начать построение требуется нажать кнопку Сreate. Также понимает файлы языка Genetics, чтоб открыть их в диалоге открытия смените тип файла на (*.gen). Save as *.ent - Сохраняет открытый файл в формате базы данных PDB. Если требуется только конвертировать в PDB, можно отключить опцию Auto create on load. Create group
Create - Начинает создавать модель молекулы из последнего открытого файла. Auto create on load - Отключает создание при открытии файла.Эта опция никак не влияет, если открывать через панель инструментов кнопкой OpenEMBL. Options group
Geometry Qality - Позволяет настроить качество геометрии. Create Wire Atoms - Включает построение проволочного соединения атомов. Show Radicals - Включает построение всей геометрии остатка. Show Sockets - Показывать соединительные кольца. Single Object - Cобирает все в один обьект. Five Angular System - Включает расширенную систему углов, разработанную Захаровым Сергеем. Setup Angles - Вызывает диалог настройки углов.
Если был открыт файл, то информация о последовательности углов будет передана для последующей с ними работы. Материал с форума лаборатории Наномир:Victoria: Что касается скрипта Дениса
Савина, то не вижу смысла осваивать способ настройки углов между аминокислотами, пока форма аминокислот остаётся НЕ верной: D, вместо -- L. Кушелев: Самое смешное, что настраивая композиционные углы, Вы работаете на другом уровне. Вам вообще не нужно знать форму аминокислот. Форму аминокислот нужно знать для более точного определения координат отдельных атомов. А для определения центров самих аминокислотных остатков их форма вообще не нужна  Скрипт Кушелева работает без форм аминокислот. Только на композиционных углах: p = #()
angx = #(-24,180,60,-45,-45); angy = #(-10.89167,83,0,15,15); angz = #(92,120,60,110,110)
dx = #(2,1.5,1.6,2,2); dy = #(0.6,1,0.8,1,0.6); dz = #(-0.45,0,-0.6,-0.15,-0.45)
kk = #()
peptide = box length:1 width:1 height:1 position:[0,0,-.5] wirecolor:[250,250,250]; Converttomesh peptide
for k = 1 to *** do(
element = Hedra family:1 scalep:100 scaleq:100 scaler:100 radius:1 pos:[0,0,0] p:0.4; Converttomesh element
attach peptide element; peptide.pivot = [0,0,0]; move peptide [dx[kk[k]],dy[kk[k]],dz[kk[k]]]
rotate peptide angx[kk[k]] [1,0,0]; rotate peptide angy[kk[k]] [0,1,0]; rotate peptide angz[kk[k]] [0,0,1]) Верхняя модель, построенная по скрипту Кушелева, не нуждается в формах аминокислот. Нижняя (с прорисовкой отдельных электронов) уже требует форму аминокислот.
Настройка композиционных углов полновесного скрипта завершена. 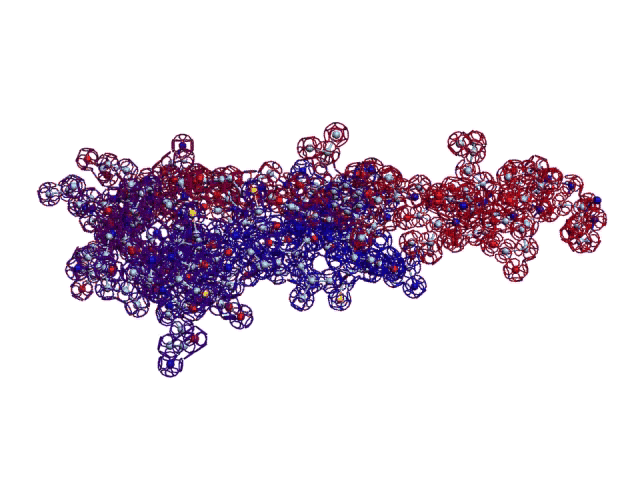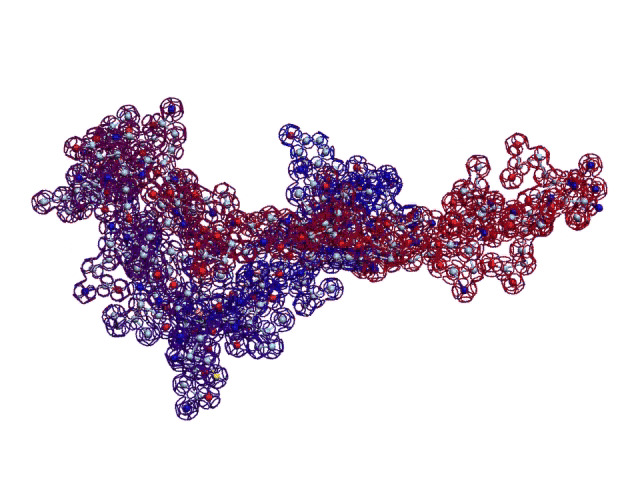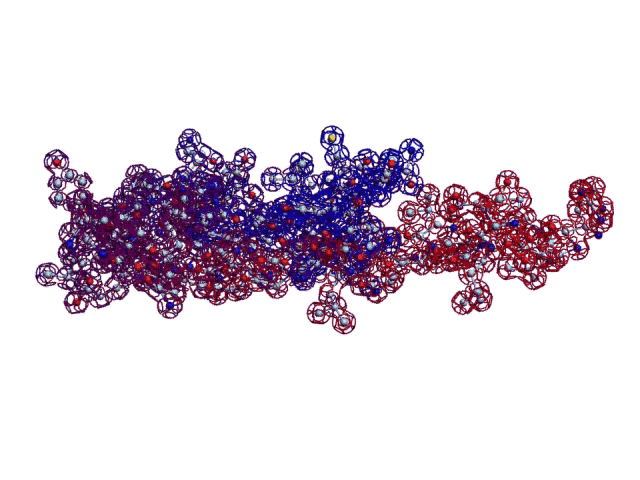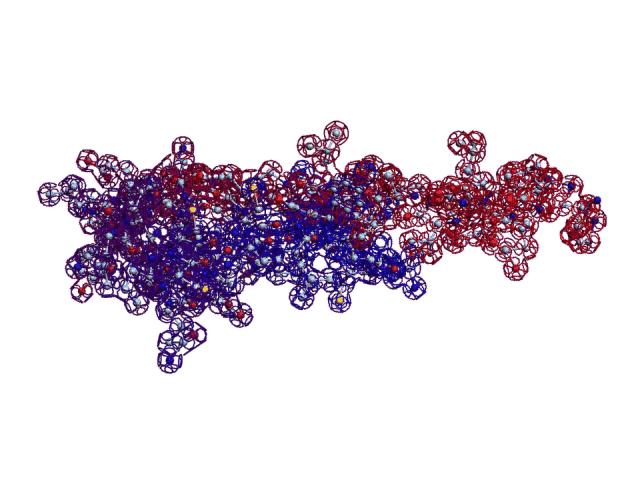 Углы в полновесном скрипте Савина-Кушелева настроены. Софт готов к получению координат атомов в стандарте PDB. Каждый желающий может заказать файл ent и посмотреть структуру белка стандартным браузером, например, http://spdbv.vital-it.ch/ Skype: 2012-01-10 [19:20:38] Кушелев Александр Юрьевич: Я автор композиционного генетического кода. Авторство числится по первой публикации. Опубликован в 1993-ем году
[19:21:13] Кушелев Александр Юрьевич: Если нужны ссылки на научные статьи, то можете дать эту: http://nanoworld88.narod.ru/forum/science/001.htm
[19:21:55] Кушелев Александр Юрьевич: Свежие научные статьи:http://nanoworld.org.ru/topic/202/ Файл с координатами атомов в стандарте PDB (Protein Data Base): Кушелев: Этот файл можно смотреть стандартным браузером. 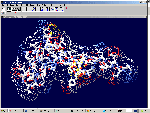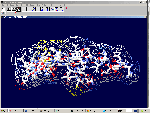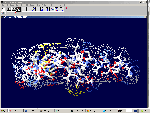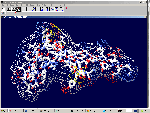 PFRMAT TS
TARGET T00xx
AUTHOR 4236-1297-6301
REMARK Predictor remarks. See DEMO:
REMARK http://www.nanoworld.org.ru/data/01/dat … 9/demo.htm
REMARK
REMARK Target sequence (56 acids)
REMARK REVEELQVGQAELGGGPGAGGLQPSALYLALQKRGIVEQCCTSICSLYQLENYCN*
REMARK
METHOD Method description
METHOD The strong correlation dependence of spatial structure
METHOD of the protein from its nucleotide sequence was
METHOD theoretically predicted by physical modelling,
METHOD experimentally discovered and statistically confirmed.
METHOD In the process of biosynthesis the third nucleotide of
METHOD the codon controls the orientation of the amino acid
METHOD forming the concrete spatial isomer that is the
METHOD conformation of the protein molecule cutting off
METHOD competition ways of the forming of 2D and 3D structures.
METHOD On this base the computer program "Pikotechnology" for
METHOD the prediction of 2D structure of the protins on their
METHOD nucleotide sequence was created.
MODEL 1
PARENT N/A 1
ATOM 1 N ARG A 1 10.504 6.874 4.374 1.00 0.50 A
ATOM 2 CA ARG A 1 9.740 6.273 4.413 1.00 0.50 A
ATOM 3 C ARG A 1 9.285 5.939 3.569 1.00 0.50 A
ATOM 4 O ARG A 1 8.721 5.994 3.174 1.00 0.50 A
ATOM 5 CB ARG A 1 8.925 6.792 4.452 1.00 0.50 A
ATOM 6 CG ARG A 1 8.345 6.773 5.159 1.00 0.50 A
ATOM 7 CD ARG A 1 8.503 6.294 5.923 1.00 0.50 A
ATOM 8 NE ARG A 1 9.472 5.753 5.827 1.00 0.50 A
ATOM 9 CZ ARG A 1 8.790 4.996 5.862 1.00 0.50 A
ATOM 10 NH1 ARG A 1 8.119 4.844 5.896 1.00 0.50 A
ATOM 11 NH2 ARG A 1 9.728 4.602 5.820 1.00 0.50 A
ATOM 12 N GLU A 2 10.213 5.511 3.641 1.00 0.50 A
ATOM 13 CA GLU A 2 9.852 5.022 2.881 1.00 0.50 A
ATOM 14 C GLU A 2 9.121 5.416 2.297 1.00 0.50 A
ATOM 15 O GLU A 2 8.451 5.382 2.135 1.00 0.50 A
ATOM 16 CB GLU A 2 9.105 4.483 3.172 1.00 0.50 A
ATOM 17 CG GLU A 2 9.318 3.589 2.905 1.00 0.50 A
ATOM 18 CD GLU A 2 8.454 3.616 2.372 1.00 0.50 A
ATOM 19 OE1 GLU A 2 7.944 4.019 2.148 1.00 0.50 A
ATOM 20 OE2 GLU A 2 8.818 2.663 2.377 1.00 0.50 A
ATOM 21 N VAL A 3 9.907 5.782 2.071 1.00 0.50 A
ATOM 22 CA VAL A 3 9.510 6.478 1.518 1.00 0.50 A
ATOM 23 C VAL A 3 8.536 6.436 1.233 1.00 0.50 A
ATOM 24 O VAL A 3 8.109 6.320 0.703 1.00 0.50 A
ATOM 25 CB VAL A 3 9.743 6.255 0.615 1.00 0.50 A
ATOM 26 CG1 VAL A 3 10.287 6.987 0.331 1.00 0.50 A
ATOM 27 CG2 VAL A 3 9.003 6.121 -0.026 1.00 0.50 A
ATOM 28 N GLU A 4 8.582 6.617 2.241 1.00 0.50 A
ATOM 29 CA GLU A 4 7.627 6.725 2.087 1.00 0.50 A
ATOM 30 C GLU A 4 7.163 6.270 1.307 1.00 0.50 A
ATOM 31 O GLU A 4 6.874 6.370 0.688 1.00 0.50 A
ATOM 32 CB GLU A 4 7.479 7.492 1.517 1.00 0.50 A
ATOM 33 CG GLU A 4 6.875 8.055 2.001 1.00 0.50 A
ATOM 34 CD GLU A 4 6.257 7.962 1.201 1.00 0.50 A
ATOM 35 OE1 GLU A 4 6.150 7.629 0.609 1.00 0.50 A
ATOM 36 OE2 GLU A 4 5.890 8.676 1.831 1.00 0.50 A
ATOM 37 N GLU A 5 7.442 5.560 1.991 1.00 0.50 A
ATOM 38 CA GLU A 5 6.914 5.017 1.379 1.00 0.50 A
ATOM 39 C GLU A 5 6.788 5.286 0.408 1.00 0.50 A
ATOM 40 O GLU A 5 6.340 5.537 -0.052 1.00 0.50 A
ATOM 41 CB GLU A 5 5.992 5.306 1.409 1.00 0.50 A
ATOM 42 CG GLU A 5 5.487 4.521 1.620 1.00 0.50 A
ATOM 43 CD GLU A 5 4.995 4.686 0.748 1.00 0.50 A
ATOM 44 OE1 GLU A 5 4.986 5.058 0.169 1.00 0.50 A
ATOM 45 OE2 GLU A 5 4.518 3.951 1.271 1.00 0.50 A
ATOM 46 N LEU A 6 7.599 4.918 0.509 1.00 0.50 A
ATOM 47 CA LEU A 6 7.916 5.190 -0.369 1.00 0.50 A
ATOM 48 C LEU A 6 7.275 5.530 -1.080 1.00 0.50 A
ATOM 49 O LEU A 6 6.961 5.375 -1.675 1.00 0.50 A
ATOM 50 CB LEU A 6 7.912 4.427 -0.962 1.00 0.50 A
ATOM 51 CG LEU A 6 8.802 4.294 -1.275 1.00 0.50 A
ATOM 52 CD1 LEU A 6 8.810 4.365 -2.233 1.00 0.50 A
ATOM 53 CD2 LEU A 6 9.078 3.398 -0.996 1.00 0.50 A
ATOM 54 N GLN A 7 7.342 6.225 -0.329 1.00 0.50 A
ATOM 55 CA GLN A 7 6.813 6.719 -0.981 1.00 0.50 A
ATOM 56 C GLN A 7 6.245 6.241 -1.673 1.00 0.50 A
ATOM 57 O GLN A 7 6.192 6.077 -2.341 1.00 0.50 A
ATOM 58 CB GLN A 7 7.347 6.883 -1.770 1.00 0.50 A
ATOM 59 CG GLN A 7 7.327 7.831 -1.898 1.00 0.50 A
ATOM 60 CD GLN A 7 6.931 7.629 -2.811 1.00 0.50 A
ATOM 61 OE1 GLN A 7 6.670 7.137 -3.214 1.00 0.50 A
ATOM 62 NE2 GLN A 7 7.164 8.621 -2.755 1.00 0.50 A
ATOM 63 N VAL A 8 5.818 6.234 -0.885 1.00 0.50 A
ATOM 64 CA VAL A 8 5.146 5.590 -1.171 1.00 0.50 A
ATOM 65 C VAL A 8 5.020 5.356 -2.151 1.00 0.50 A
ATOM 66 O VAL A 8 4.609 5.435 -2.700 1.00 0.50 A
ATOM 67 CB VAL A 8 4.323 6.082 -1.223 1.00 0.50 A
ATOM 68 CG1 VAL A 8 3.767 5.671 -0.565 1.00 0.50 A
ATOM 69 CG2 VAL A 8 3.854 6.095 -2.094 1.00 0.50 A
ATOM 70 N GLY A 9 5.735 4.920 -1.833 1.00 0.50 A
ATOM 71 CA GLY A 9 6.047 4.637 -2.711 1.00 0.50 A
ATOM 72 C GLY A 9 5.452 4.707 -3.531 1.00 0.50 A
ATOM 73 O GLY A 9 5.074 4.349 -3.985 1.00 0.50 A
ATOM 74 N GLN A 10 5.717 5.650 -3.228 1.00 0.50 A
ATOM 75 CA GLN A 10 5.270 5.854 -4.068 1.00 0.50 A
ATOM 76 C GLN A 10 4.567 5.233 -4.458 1.00 0.50 A
ATOM 77 O GLN A 10 4.437 4.772 -4.955 1.00 0.50 A
ATOM 78 CB GLN A 10 5.775 5.478 -4.801 1.00 0.50 A
ATOM 79 CG GLN A 10 5.954 6.211 -5.389 1.00 0.50 A
ATOM 80 CD GLN A 10 5.467 5.673 -6.098 1.00 0.50 A
ATOM 81 OE1 GLN A 10 5.079 5.117 -6.214 1.00 0.50 A
ATOM 82 NE2 GLN A 10 5.915 6.479 -6.534 1.00 0.50 A
ATOM 83 N ALA A 11 4.200 5.716 -3.798 1.00 0.50 A
ATOM 84 CA ALA A 11 3.386 5.183 -3.760 1.00 0.50 A
ATOM 85 C ALA A 11 3.150 4.524 -4.495 1.00 0.50 A
ATOM 86 O ALA A 11 2.732 4.406 -5.032 1.00 0.50 A
ATOM 87 CB ALA A 11 2.666 5.667 -4.185 1.00 0.50 A
ATOM 88 N GLU A 12 3.959 4.170 -3.975 1.00 0.50 A
ATOM 89 CA GLU A 12 3.768 3.405 -4.547 1.00 0.50 A
ATOM 90 C GLU A 12 3.287 3.515 -5.434 1.00 0.50 A
ATOM 91 O GLU A 12 2.691 3.414 -5.768 1.00 0.50 A
ATOM 92 CB GLU A 12 2.907 3.035 -4.311 1.00 0.50 A
ATOM 93 CG GLU A 12 3.072 2.112 -4.118 1.00 0.50 A
ATOM 94 CD GLU A 12 2.405 1.910 -4.855 1.00 0.50 A
ATOM 95 OE1 GLU A 12 2.034 2.179 -5.368 1.00 0.50 A
ATOM 96 OE2 GLU A 12 2.630 1.048 -4.357 1.00 0.50 A
ATOM 97 N LEU A 13 4.144 3.756 -5.533 1.00 0.50 A
ATOM 98 CA LEU A 13 4.010 4.154 -6.411 1.00 0.50 A
ATOM 99 C LEU A 13 3.159 3.992 -6.941 1.00 0.50 A
ATOM 100 O LEU A 13 2.888 3.663 -7.483 1.00 0.50 A
ATOM 101 CB LEU A 13 4.353 3.557 -7.090 1.00 0.50 A
ATOM 102 CG LEU A 13 5.029 4.023 -7.575 1.00 0.50 A
ATOM 103 CD1 LEU A 13 4.773 4.068 -8.500 1.00 0.50 A
ATOM 104 CD2 LEU A 13 5.859 3.518 -7.473 1.00 0.50 A
ATOM 105 N GLY A 14 2.942 4.579 -6.129 1.00 0.50 A
ATOM 106 CA GLY A 14 2.091 4.609 -6.601 1.00 0.50 A
ATOM 107 C GLY A 14 1.812 3.867 -7.235 1.00 0.50 A
ATOM 108 O GLY A 14 1.725 3.697 -7.898 1.00 0.50 A
ATOM 109 N GLY A 15 1.676 3.599 -6.391 1.00 0.50 A
ATOM 110 CA GLY A 15 1.516 2.670 -6.632 1.00 0.50 A
ATOM 111 C GLY A 15 1.348 2.394 -7.595 1.00 0.50 A
ATOM 112 O GLY A 15 0.869 2.183 -8.045 1.00 0.50 A
ATOM 113 N GLY A 16 2.326 2.683 -7.495 1.00 0.50 A
ATOM 114 CA GLY A 16 2.337 2.336 -8.404 1.00 0.50 A
ATOM 115 C GLY A 16 1.494 2.306 -8.969 1.00 0.50 A
ATOM 116 O GLY A 16 1.011 1.894 -9.238 1.00 0.50 A
ATOM 117 N PRO A 17 1.780 3.283 -8.844 1.00 0.50 A
ATOM 118 CA PRO A 17 1.059 3.392 -9.488 1.00 0.50 A
ATOM 119 C PRO A 17 0.311 2.709 -9.566 1.00 0.50 A
ATOM 120 O PRO A 17 0.051 2.200 -9.953 1.00 0.50 A
ATOM 121 CB PRO A 17 1.307 2.972 -10.323 1.00 0.50 A
ATOM 122 CG PRO A 17 1.222 3.657 -10.986 1.00 0.50 A
ATOM 123 CD PRO A 17 0.561 3.037 -11.442 1.00 0.50 A
ATOM 124 OE1 PRO A 17 0.197 2.457 -11.377 1.00 0.50 A
ATOM 125 OE2 PRO A 17 0.774 3.819 -12.063 1.00 0.50 A
ATOM 126 N GLY A 18 0.216 3.210 -8.677 1.00 0.50 A
ATOM 127 CA GLY A 18 -0.609 2.695 -8.677 1.00 0.50 A
ATOM 128 C GLY A 18 -0.690 1.827 -9.197 1.00 0.50 A
ATOM 129 O GLY A 18 -0.966 1.528 -9.755 1.00 0.50 A
ATOM 130 N ALA A 19 -0.243 1.673 -8.436 1.00 0.50 A
ATOM 131 CA ALA A 19 0.044 0.781 -8.696 1.00 0.50 A
ATOM 132 C ALA A 19 -0.369 0.302 -9.491 1.00 0.50 A
ATOM 133 O ALA A 19 -0.795 -0.206 -9.682 1.00 0.50 A
ATOM 134 CB ALA A 19 -0.489 0.123 -8.230 1.00 0.50 A
ATOM 135 N GLY A 20 0.196 1.087 -9.830 1.00 0.50 A
ATOM 136 CA GLY A 20 -0.029 0.655 -10.673 1.00 0.50 A
ATOM 137 C GLY A 20 -0.860 0.079 -10.766 1.00 0.50 A
ATOM 138 O GLY A 20 -1.094 -0.567 -10.826 1.00 0.50 A
ATOM 139 N GLY A 21 -1.141 1.060 -10.660 1.00 0.50 A
ATOM 140 CA GLY A 21 -2.002 0.651 -10.861 1.00 0.50 A
ATOM 141 C GLY A 21 -2.180 -0.329 -10.666 1.00 0.50 A
ATOM 142 O GLY A 21 -2.261 -0.953 -10.948 1.00 0.50 A
ATOM 143 N LEU A 22 -2.242 0.138 -9.904 1.00 0.50 A
ATOM 144 CA LEU A 22 -2.261 -0.649 -9.333 1.00 0.50 A
ATOM 145 C LEU A 22 -2.415 -1.576 -9.716 1.00 0.50 A
ATOM 146 O LEU A 22 -2.869 -2.093 -9.775 1.00 0.50 A
ATOM 147 CB LEU A 22 -3.165 -0.802 -9.027 1.00 0.50 A
ATOM 148 CG LEU A 22 -3.161 -0.773 -8.074 1.00 0.50 A
ATOM 149 CD1 LEU A 22 -3.471 -1.617 -7.735 1.00 0.50 A
ATOM 150 CD2 LEU A 22 -3.764 -0.052 -7.804 1.00 0.50 A
ATOM 151 N GLN A 23 -1.492 -1.241 -10.011 1.00 0.50 A
ATOM 152 CA GLN A 23 -1.456 -2.159 -10.332 1.00 0.50 A
ATOM 153 C GLN A 23 -2.302 -2.677 -10.549 1.00 0.50 A
ATOM 154 O GLN A 23 -2.723 -3.183 -10.339 1.00 0.50 A
ATOM 155 CB GLN A 23 -1.476 -2.764 -9.577 1.00 0.50 A
ATOM 156 CG GLN A 23 -0.698 -3.313 -9.673 1.00 0.50 A
ATOM 157 CD GLN A 23 -1.340 -4.099 -9.688 1.00 0.50 A
ATOM 158 OE1 GLN A 23 -2.002 -4.284 -9.722 1.00 0.50 A
ATOM 159 NE2 GLN A 23 -0.383 -4.446 -9.619 1.00 0.50 A
ATOM 160 N PRO A 24 -2.172 -1.939 -11.248 1.00 0.50 A
ATOM 161 CA PRO A 24 -2.923 -2.420 -11.638 1.00 0.50 A
ATOM 162 C PRO A 24 -3.554 -2.970 -11.064 1.00 0.50 A
ATOM 163 O PRO A 24 -3.743 -3.611 -10.890 1.00 0.50 A
ATOM 164 CB PRO A 24 -2.639 -3.308 -11.891 1.00 0.50 A
ATOM 165 CG PRO A 24 -2.854 -3.389 -12.820 1.00 0.50 A
ATOM 166 CD PRO A 24 -3.423 -4.184 -12.548 1.00 0.50 A
ATOM 167 OE1 PRO A 24 -3.688 -4.531 -12.015 1.00 0.50 A
ATOM 168 OE2 PRO A 24 -3.359 -4.148 -13.565 1.00 0.50 A
ATOM 169 N SER A 25 -3.764 -2.103 -10.984 1.00 0.50 A
ATOM 170 CA SER A 25 -4.365 -2.240 -10.231 1.00 0.50 A
ATOM 171 C SER A 25 -4.688 -3.168 -9.974 1.00 0.50 A
ATOM 172 O SER A 25 -5.229 -3.596 -9.976 1.00 0.50 A
ATOM 173 CB SER A 25 -5.247 -2.069 -10.569 1.00 0.50 A
ATOM 174 OG SER A 25 -5.862 -2.793 -10.490 1.00 0.50 A
ATOM 175 N ALA A 26 -3.852 -3.053 -9.673 1.00 0.50 A
ATOM 176 CA ALA A 26 -3.724 -3.991 -9.446 1.00 0.50 A
ATOM 177 C ALA A 26 -4.507 -4.637 -9.406 1.00 0.50 A
ATOM 178 O ALA A 26 -4.918 -4.998 -8.985 1.00 0.50 A
ATOM 179 CB ALA A 26 -3.794 -4.103 -8.489 1.00 0.50 A
ATOM 180 N LEU A 27 -4.207 -4.593 -10.458 1.00 0.50 A
ATOM 181 CA LEU A 27 -5.147 -4.844 -10.425 1.00 0.50 A
ATOM 182 C LEU A 27 -5.883 -4.161 -10.575 1.00 0.50 A
ATOM 183 O LEU A 27 -6.378 -3.790 -10.268 1.00 0.50 A
ATOM 184 CB LEU A 27 -5.472 -4.753 -9.519 1.00 0.50 A
ATOM 185 CG LEU A 27 -5.788 -5.607 -9.233 1.00 0.50 A
ATOM 186 CD1 LEU A 27 -6.719 -5.534 -9.007 1.00 0.50 A
ATOM 187 CD2 LEU A 27 -5.263 -5.863 -8.449 1.00 0.50 A
ATOM 188 N TYR A 28 -5.573 -4.492 -11.571 1.00 0.50 A
ATOM 189 CA TYR A 28 -5.947 -3.598 -11.656 1.00 0.50 A
ATOM 190 C TYR A 28 -5.350 -2.778 -11.710 1.00 0.50 A
ATOM 191 O TYR A 28 -5.123 -2.223 -11.368 1.00 0.50 A
ATOM 192 CB TYR A 28 -6.098 -3.228 -10.776 1.00 0.50 A
ATOM 193 CD2 TYR A 28 -6.914 -2.876 -10.259 1.00 0.50 A
ATOM 194 CE2 TYR A 28 -7.465 -2.649 -9.913 1.00 0.50 A
ATOM 195 CZ TYR A 28 -8.088 -2.659 -10.212 1.00 0.50 A
ATOM 196 CE1 TYR A 28 -8.159 -2.901 -10.853 1.00 0.50 A
ATOM 197 CD1 TYR A 28 -7.609 -3.128 -11.199 1.00 0.50 A
ATOM 198 CG TYR A 28 -6.984 -3.118 -10.902 1.00 0.50 A
ATOM 199 OH TYR A 28 -8.977 -2.551 -10.336 1.00 0.50 A
ATOM 200 N LEU A 29 -5.365 -3.185 -12.509 1.00 0.50 A
ATOM 201 CA LEU A 29 -4.629 -2.663 -12.873 1.00 0.50 A
ATOM 202 C LEU A 29 -4.297 -1.822 -12.410 1.00 0.50 A
ATOM 203 O LEU A 29 -4.281 -1.132 -12.439 1.00 0.50 A
ATOM 204 CB LEU A 29 -4.978 -1.995 -13.479 1.00 0.50 A
ATOM 205 CG LEU A 29 -4.588 -2.145 -14.336 1.00 0.50 A
ATOM 206 CD1 LEU A 29 -4.101 -1.360 -14.599 1.00 0.50 A
ATOM 207 CD2 LEU A 29 -5.319 -2.321 -14.961 1.00 0.50 A
ATOM 208 N ALA A 30 -4.047 -2.638 -11.842 1.00 0.50 A
ATOM 209 CA ALA A 30 -3.573 -1.937 -11.361 1.00 0.50 A
ATOM 210 C ALA A 30 -3.875 -0.968 -11.404 1.00 0.50 A
ATOM 211 O ALA A 30 -3.731 -0.344 -11.662 1.00 0.50 A
ATOM 212 CB ALA A 30 -2.877 -1.575 -11.926 1.00 0.50 A
ATOM 213 N LEU A 31 -4.403 -1.417 -10.835 1.00 0.50 A
ATOM 214 CA LEU A 31 -5.061 -0.702 -10.765 1.00 0.50 A
ATOM 215 C LEU A 31 -4.863 0.234 -11.102 1.00 0.50 A
ATOM 216 O LEU A 31 -4.719 0.882 -10.912 1.00 0.50 A
ATOM 217 CB LEU A 31 -4.949 -0.246 -9.920 1.00 0.50 A
ATOM 218 CG LEU A 31 -5.768 -0.319 -9.437 1.00 0.50 A
ATOM 219 CD1 LEU A 31 -6.105 0.563 -9.261 1.00 0.50 A
ATOM 220 CD2 LEU A 31 -5.571 -0.784 -8.600 1.00 0.50 A
ATOM 221 N GLN A 32 -4.952 -0.398 -11.903 1.00 0.50 A
ATOM 222 CA GLN A 32 -4.892 0.458 -12.361 1.00 0.50 A
ATOM 223 C GLN A 32 -4.434 1.256 -11.932 1.00 0.50 A
ATOM 224 O GLN A 32 -4.536 1.870 -11.632 1.00 0.50 A
ATOM 225 CB GLN A 32 -5.668 0.993 -12.144 1.00 0.50 A
ATOM 226 CG GLN A 32 -6.057 1.228 -12.986 1.00 0.50 A
ATOM 227 CD GLN A 32 -5.925 2.193 -12.700 1.00 0.50 A
ATOM 228 OE1 GLN A 32 -5.636 2.606 -12.232 1.00 0.50 A
ATOM 229 NE2 GLN A 32 -6.494 2.167 -13.546 1.00 0.50 A
ATOM 230 N LYS A 33 -3.727 0.554 -12.172 1.00 0.50 A
ATOM 231 CA LYS A 33 -3.155 1.291 -11.896 1.00 0.50 A
ATOM 232 C LYS A 33 -3.486 2.006 -11.255 1.00 0.50 A
ATOM 233 O LYS A 33 -3.691 2.663 -11.202 1.00 0.50 A
ATOM 234 CB LYS A 33 -3.260 2.027 -12.514 1.00 0.50 A
ATOM 235 CG LYS A 33 -2.583 2.352 -13.036 1.00 0.50 A
ATOM 236 CD LYS A 33 -1.747 1.980 -13.059 1.00 0.50 A
ATOM 237 CE LYS A 33 -1.506 1.253 -12.559 1.00 0.50 A
ATOM 238 NZ LYS A 33 -2.077 0.823 -11.990 1.00 0.50 A
ATOM 239 N ARG A 34 -3.379 1.123 -10.746 1.00 0.50 A
ATOM 240 CA ARG A 34 -3.572 1.729 -10.009 1.00 0.50 A
ATOM 241 C ARG A 34 -4.090 2.590 -10.154 1.00 0.50 A
ATOM 242 O ARG A 34 -4.040 3.278 -10.188 1.00 0.50 A
ATOM 243 CB ARG A 34 -2.852 2.368 -9.922 1.00 0.50 A
ATOM 244 CG ARG A 34 -2.319 2.434 -9.182 1.00 0.50 A
ATOM 245 CD ARG A 34 -2.386 1.867 -8.466 1.00 0.50 A
ATOM 246 NE ARG A 34 -3.205 1.136 -8.647 1.00 0.50 A
ATOM 247 CZ ARG A 34 -3.554 1.678 -7.857 1.00 0.50 A
ATOM 248 NH1 ARG A 34 -3.423 2.208 -7.437 1.00 0.50 A
ATOM 249 NH2 ARG A 34 -4.219 0.939 -8.078 1.00 0.50 A
ATOM 250 N GLY A 35 -4.724 1.794 -10.275 1.00 0.50 A
ATOM 251 CA GLY A 35 -5.358 2.531 -10.314 1.00 0.50 A
ATOM 252 C GLY A 35 -5.069 3.462 -10.599 1.00 0.50 A
ATOM 253 O GLY A 35 -4.940 4.103 -10.378 1.00 0.50 A
ATOM 254 N ILE A 36 -5.035 2.838 -11.411 1.00 0.50 A
ATOM 255 CA ILE A 36 -4.864 3.695 -11.840 1.00 0.50 A
ATOM 256 C ILE A 36 -4.463 4.473 -11.325 1.00 0.50 A
ATOM 257 O ILE A 36 -4.595 5.088 -11.041 1.00 0.50 A
ATOM 258 CB ILE A 36 -5.701 4.165 -11.821 1.00 0.50 A
ATOM 259 CG1 ILE A 36 -5.923 4.274 -12.744 1.00 0.50 A
ATOM 260 CG2 ILE A 36 -5.720 5.044 -11.368 1.00 0.50 A
ATOM 261 CD1 ILE A 36 -5.247 3.879 -13.261 1.00 0.50 A
ATOM 262 N VAL A 37 -3.812 3.896 -11.541 1.00 0.50 A
ATOM 263 CA VAL A 37 -3.148 4.312 -10.963 1.00 0.50 A
ATOM 264 C VAL A 37 -3.300 5.232 -10.560 1.00 0.50 A
ATOM 265 O VAL A 37 -3.117 5.896 -10.589 1.00 0.50 A
ATOM 266 CB VAL A 37 -2.511 4.720 -11.554 1.00 0.50 A
ATOM 267 CG1 VAL A 37 -1.698 4.253 -11.373 1.00 0.50 A
ATOM 268 CG2 VAL A 37 -2.364 5.693 -11.464 1.00 0.50 A
ATOM 269 N GLU A 38 -3.940 4.563 -10.120 1.00 0.50 A
ATOM 270 CA GLU A 38 -4.100 5.348 -9.568 1.00 0.50 A
ATOM 271 C GLU A 38 -3.931 6.286 -9.920 1.00 0.50 A
ATOM 272 O GLU A 38 -3.511 6.833 -9.885 1.00 0.50 A
ATOM 273 CB GLU A 38 -3.296 5.564 -9.076 1.00 0.50 A
ATOM 274 CG GLU A 38 -3.558 5.581 -8.156 1.00 0.50 A
ATOM 275 CD GLU A 38 -3.226 6.540 -8.151 1.00 0.50 A
ATOM 276 OE1 GLU A 38 -3.004 7.060 -8.542 1.00 0.50 A
ATOM 277 OE2 GLU A 38 -3.430 6.248 -7.195 1.00 0.50 A
ATOM 278 N GLN A 39 -4.743 5.888 -10.402 1.00 0.50 A
ATOM 279 CA GLN A 39 -4.772 6.803 -10.734 1.00 0.50 A
ATOM 280 C GLN A 39 -3.936 7.379 -10.771 1.00 0.50 A
ATOM 281 O GLN A 39 -3.607 7.912 -10.481 1.00 0.50 A
ATOM 282 CB GLN A 39 -4.957 7.404 -10.001 1.00 0.50 A
ATOM 283 CG GLN A 39 -5.732 7.899 -10.268 1.00 0.50 A
ATOM 284 CD GLN A 39 -5.157 8.728 -10.153 1.00 0.50 A
ATOM 285 OE1 GLN A 39 -4.518 8.959 -10.047 1.00 0.50 A
ATOM 286 NE2 GLN A 39 -6.128 9.007 -10.297 1.00 0.50 A
ATOM 287 N CYS A 40 -3.931 6.782 -11.689 1.00 0.50 A
ATOM 288 CA CYS A 40 -3.010 7.022 -11.481 1.00 0.50 A
ATOM 289 C CYS A 40 -2.385 6.369 -11.017 1.00 0.50 A
ATOM 290 O CYS A 40 -2.066 6.221 -10.424 1.00 0.50 A
ATOM 291 CB CYS A 40 -2.974 7.427 -10.604 1.00 0.50 A
ATOM 292 SG CYS A 40 -2.614 8.304 -10.701 1.00 0.50 A
ATOM 293 N CYS A 41 -2.573 6.035 -11.969 1.00 0.50 A
ATOM 294 CA CYS A 41 -1.884 5.405 -11.693 1.00 0.50 A
ATOM 295 C CYS A 41 -1.724 5.190 -10.714 1.00 0.50 A
ATOM 296 O CYS A 41 -1.297 5.284 -10.179 1.00 0.50 A
ATOM 297 CB CYS A 41 -1.052 5.878 -11.557 1.00 0.50 A
ATOM 298 SG CYS A 41 -0.402 5.504 -12.147 1.00 0.50 A
ATOM 299 N THR A 42 -2.625 4.794 -11.001 1.00 0.50 A
ATOM 300 CA THR A 42 -2.543 4.417 -10.107 1.00 0.50 A
ATOM 301 C THR A 42 -1.982 4.868 -9.392 1.00 0.50 A
ATOM 302 O THR A 42 -1.393 4.833 -9.033 1.00 0.50 A
ATOM 303 CB THR A 42 -1.856 3.750 -10.171 1.00 0.50 A
ATOM 304 OG1 THR A 42 -2.309 2.934 -9.970 1.00 0.50 A
ATOM 305 CG2 THR A 42 -1.070 3.848 -9.579 1.00 0.50 A
ATOM 306 N SER A 43 -2.811 5.441 -9.577 1.00 0.50 A
ATOM 307 CA SER A 43 -2.442 5.910 -8.808 1.00 0.50 A
ATOM 308 C SER A 43 -1.453 5.861 -8.584 1.00 0.50 A
ATOM 309 O SER A 43 -0.986 5.597 -8.150 1.00 0.50 A
ATOM 310 CB SER A 43 -2.601 5.336 -8.055 1.00 0.50 A
ATOM 311 OG SER A 43 -1.822 5.065 -7.578 1.00 0.50 A
ATOM 312 N ILE A 44 -1.576 6.407 -9.443 1.00 0.50 A
ATOM 313 CA ILE A 44 -0.626 6.545 -9.281 1.00 0.50 A
ATOM 314 C ILE A 44 -0.077 5.877 -8.748 1.00 0.50 A
ATOM 315 O ILE A 44 0.238 5.762 -8.145 1.00 0.50 A
ATOM 316 CB ILE A 44 -0.576 7.132 -8.523 1.00 0.50 A
ATOM 317 CG1 ILE A 44 -0.157 7.918 -8.869 1.00 0.50 A
ATOM 318 CG2 ILE A 44 -0.080 6.811 -7.731 1.00 0.50 A
ATOM 319 CD1 ILE A 44 0.017 7.785 -9.781 1.00 0.50 A
ATOM 320 N CYS A 45 -0.337 5.457 -9.646 1.00 0.50 A
ATOM 321 CA CYS A 45 0.271 4.779 -9.304 1.00 0.50 A
ATOM 322 C CYS A 45 0.432 4.668 -8.307 1.00 0.50 A
ATOM 323 O CYS A 45 0.884 4.769 -7.795 1.00 0.50 A
ATOM 324 CB CYS A 45 1.162 5.149 -9.241 1.00 0.50 A
ATOM 325 SG CYS A 45 1.738 4.622 -9.788 1.00 0.50 A
ATOM 326 N SER A 46 -0.348 4.286 -8.524 1.00 0.50 A
ATOM 327 CA SER A 46 -0.633 4.168 -7.600 1.00 0.50 A
ATOM 328 C SER A 46 0.018 4.274 -6.828 1.00 0.50 A
ATOM 329 O SER A 46 0.379 3.937 -6.346 1.00 0.50 A
ATOM 330 CB SER A 46 -0.607 3.222 -7.441 1.00 0.50 A
ATOM 331 OG SER A 46 -0.037 2.943 -6.729 1.00 0.50 A
ATOM 332 N LEU A 47 -0.153 5.192 -7.251 1.00 0.50 A
ATOM 333 CA LEU A 47 0.369 5.452 -6.472 1.00 0.50 A
ATOM 334 C LEU A 47 1.017 4.806 -6.031 1.00 0.50 A
ATOM 335 O LEU A 47 1.125 4.406 -5.479 1.00 0.50 A
ATOM 336 CB LEU A 47 -0.127 5.247 -5.669 1.00 0.50 A
ATOM 337 CG LEU A 47 -0.253 6.059 -5.186 1.00 0.50 A
ATOM 338 CD1 LEU A 47 0.150 5.972 -4.319 1.00 0.50 A
ATOM 339 CD2 LEU A 47 -1.217 6.201 -5.103 1.00 0.50 A
ATOM 340 N TYR A 48 1.387 5.079 -6.947 1.00 0.50 A
ATOM 341 CA TYR A 48 2.143 4.577 -6.594 1.00 0.50 A
ATOM 342 C TYR A 48 2.018 3.859 -5.887 1.00 0.50 A
ATOM 343 O TYR A 48 2.108 3.704 -5.220 1.00 0.50 A
ATOM 344 CB TYR A 48 2.505 5.040 -5.827 1.00 0.50 A
ATOM 345 CD2 TYR A 48 3.418 5.442 -5.577 1.00 0.50 A
ATOM 346 CE2 TYR A 48 4.029 5.718 -5.417 1.00 0.50 A
ATOM 347 CZ TYR A 48 4.550 5.687 -5.870 1.00 0.50 A
ATOM 348 CE1 TYR A 48 4.457 5.387 -6.482 1.00 0.50 A
ATOM 349 CD1 TYR A 48 3.847 5.111 -6.642 1.00 0.50 A
ATOM 350 CG TYR A 48 3.324 5.139 -6.191 1.00 0.50 A
ATOM 351 OH TYR A 48 5.371 5.790 -6.235 1.00 0.50 A
ATOM 352 N GLN A 49 1.618 3.531 -6.772 1.00 0.50 A
ATOM 353 CA GLN A 49 1.545 2.722 -6.235 1.00 0.50 A
ATOM 354 C GLN A 49 1.494 2.763 -5.221 1.00 0.50 A
ATOM 355 O GLN A 49 1.841 2.648 -4.635 1.00 0.50 A
ATOM 356 CB GLN A 49 2.430 2.402 -6.015 1.00 0.50 A
ATOM 357 CG GLN A 49 2.457 1.497 -6.325 1.00 0.50 A
ATOM 358 CD GLN A 49 2.665 1.246 -5.364 1.00 0.50 A
ATOM 359 OE1 GLN A 49 2.699 1.478 -4.717 1.00 0.50 A
ATOM 360 NE2 GLN A 49 2.792 0.429 -5.961 1.00 0.50 A
ATOM 361 N LEU A 50 0.692 2.967 -5.565 1.00 0.50 A
ATOM 362 CA LEU A 50 0.327 3.285 -4.720 1.00 0.50 A
ATOM 363 C LEU A 50 0.796 3.100 -3.839 1.00 0.50 A
ATOM 364 O LEU A 50 0.774 2.730 -3.257 1.00 0.50 A
ATOM 365 CB LEU A 50 -0.268 2.617 -4.357 1.00 0.50 A
ATOM 366 CG LEU A 50 -1.130 3.014 -4.262 1.00 0.50 A
ATOM 367 CD1 LEU A 50 -1.389 2.980 -3.337 1.00 0.50 A
ATOM 368 CD2 LEU A 50 -1.751 2.496 -4.811 1.00 0.50 A
ATOM 369 N GLU A 51 1.354 3.767 -4.381 1.00 0.50 A
ATOM 370 CA GLU A 51 1.838 3.778 -3.537 1.00 0.50 A
ATOM 371 C GLU A 51 1.807 2.988 -2.899 1.00 0.50 A
ATOM 372 O GLU A 51 1.555 2.759 -2.299 1.00 0.50 A
ATOM 373 CB GLU A 51 1.272 4.149 -2.847 1.00 0.50 A
ATOM 374 CG GLU A 51 1.770 4.877 -2.475 1.00 0.50 A
ATOM 375 CD GLU A 51 1.689 4.389 -1.589 1.00 0.50 A
ATOM 376 OE1 GLU A 51 1.499 3.801 -1.285 1.00 0.50 A
ATOM 377 OE2 GLU A 51 2.068 5.321 -1.426 1.00 0.50 A
ATOM 378 N ASN A 52 2.340 2.724 -3.734 1.00 0.50 A
ATOM 379 CA ASN A 52 2.494 1.944 -3.172 1.00 0.50 A
ATOM 380 C ASN A 52 1.841 1.682 -2.440 1.00 0.50 A
ATOM 381 O ASN A 52 1.738 1.663 -1.757 1.00 0.50 A
ATOM 382 CB ASN A 52 3.008 2.204 -2.391 1.00 0.50 A
ATOM 383 CG ASN A 52 3.643 1.521 -2.793 1.00 0.50 A
ATOM 384 OD1 ASN A 52 3.737 1.079 -3.315 1.00 0.50 A
ATOM 385 ND2 ASN A 52 4.051 1.898 -1.940 1.00 0.50 A
ATOM 386 N TYR A 53 1.405 1.516 -3.353 1.00 0.50 A
ATOM 387 CA TYR A 53 0.740 1.115 -2.765 1.00 0.50 A
ATOM 388 C TYR A 53 0.644 1.409 -1.797 1.00 0.50 A
ATOM 389 O TYR A 53 0.758 1.230 -1.141 1.00 0.50 A
ATOM 390 CB TYR A 53 1.124 0.346 -2.322 1.00 0.50 A
ATOM 391 CD2 TYR A 53 0.904 -0.656 -2.273 1.00 0.50 A
ATOM 392 CE2 TYR A 53 0.758 -1.328 -2.250 1.00 0.50 A
ATOM 393 CZ TYR A 53 0.219 -1.577 -2.605 1.00 0.50 A
ATOM 394 CE1 TYR A 53 -0.167 -1.153 -2.985 1.00 0.50 A
ATOM 395 CD1 TYR A 53 -0.022 -0.480 -3.008 1.00 0.50 A
ATOM 396 CG TYR A 53 0.515 -0.229 -2.654 1.00 0.50 A
ATOM 397 OH TYR A 53 -0.387 -2.157 -2.939 1.00 0.50 A
ATOM 398 N CYS A 54 0.323 2.165 -2.411 1.00 0.50 A
ATOM 399 CA CYS A 54 0.064 2.521 -1.543 1.00 0.50 A
ATOM 400 C CYS A 54 0.539 2.236 -0.691 1.00 0.50 A
ATOM 401 O CYS A 54 0.483 1.861 -0.114 1.00 0.50 A
ATOM 402 CB CYS A 54 -0.619 1.959 -1.156 1.00 0.50 A
ATOM 403 SG CYS A 54 -1.392 2.497 -1.007 1.00 0.50 A
ATOM 404 N ASN A 55 1.176 2.809 -1.254 1.00 0.50 A
ATOM 405 CA ASN A 55 1.698 2.715 -0.438 1.00 0.50 A
ATOM 406 C ASN A 55 1.563 1.928 0.188 1.00 0.50 A
ATOM 407 O ASN A 55 1.306 1.732 0.798 1.00 0.50 A
ATOM 408 CB ASN A 55 1.240 3.162 0.291 1.00 0.50 A
ATOM 409 CG ASN A 55 2.111 3.686 0.290 1.00 0.50 A
ATOM 410 OD1 ASN A 55 2.726 3.753 -0.014 1.00 0.50 A
ATOM 411 ND2 ASN A 55 1.503 4.045 1.024 1.00 0.50 A
TER
END
Обсуждение на форуме лаборатории Наномир.
В Манчестере запущен "голый сканер" 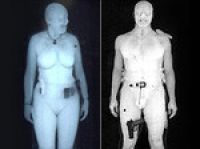
Приглашение к сотрудничеству для людей умеющих самостоятельно мыслить; не просто умных, а мудрых, которые чувствуют, где истина
Лаборатория Наномир готова к любому взаимовыгодному сотрудничеству. У нас есть сторонники как явные, которые помогают морально и материально, есть очень много пассивных наблюдателей,
есть и ярые противники, которые используют любые методы и средства (аморальные и просто преступные), чтобы уничтожить работу лаборатории и дискредитировать ее. В одиночку внедрить технологии, выводящие цивилизацию на новый уровень, невозможно. Благодаря поддержке множества заинтересованных людей проделана огромная работа. Ознакомиться с её результатами можно изучив материал рассылки "Новости лаборатории Наномир". Люди
науки могут изучить научные труды.
Вклад каждого не останется незамеченным в случае успеха в реализации научных проектов. Результаты совместной деятельности принадлежат участникам проекта пропорционально коэффициентам творческого и финансового участия. В этом году были куплены рубиновые шарики для эксперимента на сумму ~1000 долл. В результате было сделано научное открытие, проверена защита диэлектрических резонаторов от
перенапряжения. В этом же году, вероятно, можно будет создать микроволновую энергетику, т.к. удалось найти сырьё (рубин #8), из которого сделаны рубиновые шарики для эксперимента в Дубне. 28 сентября начался эксперимент по созданию "эликсира вечной молодости". Благодаря первому взносу (в размере 500 долларов) Золдракса и поддержке других соинвесторов. Продолжаются переговоры с потенциальными инвесторами по поводу финансирования этого проекта. Созданы первые версии пикотехнологии, с помощью
которой Александр Кушелев и Виктория Соколик сделали более10 научных открытий.
Сотрудничество может быть различным: - участие в научных дискуссиях на форуме (конструктивное) - совместное создание коммерческого продукта - поиск инвесторов - выступить менеджером по продаже готовых коммерческих продуктов - конструктивные предложения по продвижению идей лаборатории Наномир - содействие в проведении экспериментов и т.п.
- написание совместных научных
статей и т.п. - материальный вклад (денежный или обеспечение оборудованием и материалами)
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир. +7-926-5101703 begin_of_the_skype_highlighting +7-926-5101703 end_of_the_skype_highlighting, +7-903-2003424 begin_of_the_skype_highlighting +7-903-2003424 end_of_the_skype_highlighting, +7-916-8265031 begin_of_the_skype_highlighting +7-916-8265031 end_of_the_skype_highlighting, Skype: Kushelev2009, mail: kushelev2011@yandex.ru
веб-мани: WM-кошелек R426964799301
|