Выпуск 270
Лаборатория Наномир
Когда реальность открывает тайны,
уходят в тень и меркнут чудеса ...
Пикософт создан!
Кушелев: В лаборатории Наномир создана коммерческая версия пикософта, которая может выдавать клиентам структуры белков в стандарте PDB и в дополнительном стандарте PIKO 3D лаборатории Наномир, т.е. в виде файлов, которые можно открывать в программе 3DS Max.
В качестве примера приведена пикотехнологическая модель инсулина.
Файл в стандарте PDB, OCINSAA.ent :
PFRMAT TS
TARGET T00xx
AUTHOR 4236-1297-6301
REMARK Predictor remarks. See DEMO:
REMARK http://www.nanoworld.org.ru/data/01/dat … 9/demo.htm
REMARK
REMARK Target sequence (56 acids)
REMARK REVEELQVGQAELGGGPGAGGLQPSALYLALQKRGIVEQCCTSICSLYQLENYCN*
REMARK
METHOD Method description
METHOD The strong correlation dependence of spatial structure
METHOD of the protein from its nucleotide sequence was
METHOD theoretically predicted by physical modelling,
METHOD experimentally discovered and statistically confirmed.
METHOD In the process of biosynthesis the third nucleotide of
METHOD the codon controls the orientation of the amino acid
METHOD forming the concrete spatial isomer that is the
METHOD conformation of the protein molecule cutting off
METHOD competition ways of the forming of 2D and 3D structures.
METHOD On this base the computer program "Pikotechnology" for
METHOD the prediction of 2D structure of the protins on their
METHOD nucleotide sequence was created.
MODEL 1
PARENT N/A 1
ATOM 1 N ARG A 1 -13.10
12.29 7.33 1.00 0.50 A
ATOM 2 CA ARG A 1 -13.52 11.19 6.90 1.00 0.50 A
ATOM 3 C ARG A 1 -13.50 11.02 5.96 1.00 0.50 A
...
ATOM 405 CG ASN A 55 2.80 3.62 0.70 1.00 0.50 A
ATOM
406 OD1 ASN A 55 3.59 3.60 0.44 1.00 0.50 A
ATOM 407 ND2 ASN A 55 1.98 4.11 1.21 1.00 0.50 A
TER
END
Так же доступны: вторичная структура в стандарте SS:
PFRMAT SS
TARGET T00xx
AUTHOR 4236-1297-6301
REMARK Predictor remarks. See DEMO:
REMARK http://www.nanoworld.org.ru/data/01/dat … 9/demo.htm
REMARK
METHOD Method description
METHOD The strong correlation dependence of spatial structure
METHOD of the protein from its nucleotide sequence was
METHOD theoretically predicted by physical modelling,
METHOD experimentally discovered and statistically confirmed.
METHOD In the process of biosynthesis the third nucleotide of
METHOD the codon controls the orientation of the amino acid
METHOD forming the concrete spatial isomer that is the
METHOD conformation of the protein molecule cutting off
METHOD competition ways of the forming of 2D and 3D structures.
METHOD On this base the computer program "Pikotechnology" for
METHOD the prediction of 2D structure of the protins on their
METHOD nucleotide sequence was created.
MODEL 1
PARENT N/A 1
R H 1.00
E H 1.00
V H 1.00
...
* H 1.00
END
и в стандарте PIKO 2D:
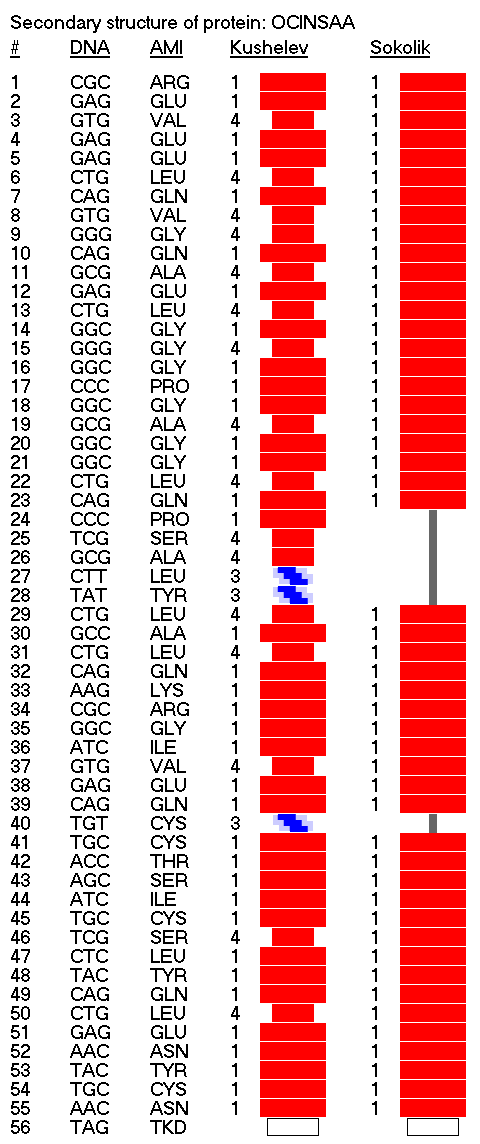
Композиционный код:
1,1,4,1,1,4,1,4,4,1,4,1,4,1,4,1,1,1,4,1,1,4,1,1,4,4,3,3,4,1,4,1,1,1,1,1,4,1,1,3,1,1,1,1,1,4,1,1,1,4,1,1,1,1,1
Входными данными для программы PikoSoft являются стандартные файлы из GenBank:
ID OCINSAA standard; RNA; MAM; 250 BP.
XX
AC M61153;
XX
DT 12-APR-1991 (Rel. 28, Created)
DT 15-APR-1994 (Rel. 39, Last updated, Version 2)
XX
DE Oryctolagus cuniculus insulin mRNA, partial cds.
XX
KW insulin.
XX
OS Oryctolagus cuniculus (rabbit)
OC Eukaryota; Animalia; Metazoa; Chordata; Vertebrata; Mammalia;
OC Theria; Eutheria; Lagomorpha; Leporidae.
XX
RN [1]
RP 1-250
RA Giddings S.J., Carnaghi L., Devaskar S.;
RT "Nucleotide sequence of
a cDNA encoding rabbit preproinsulin";
RL Unpublished.
XX
CC NCBI gi: 165444
XX
FH Key Location/Qualifiers
FH
FT source
1..250
FT /organism="Oryctolagus cuniculus"
FT CDS <1..168
FT /note="NCBI gi: 470321"
FT /product="insulin"
FT /codon_start=1
XX
SQ Sequence 250 BP; 36 A; 105 C; 78 G; 31 T; 0 other;
cgcgaggtgg aggagctgca ggtggggcag gcggagctgg gcgggggccc cggcgcgggc
ggcctgcagc cctcggcgct ttatctggcc ctgcagaagc gcggcatcgt ggagcagtgt
tgcaccagca tctgctcgct ctaccagctg gagaactact gcaactaggg gtgcccccca
cccacccctg cccgcgcccc ccacgccccc cgccctcgcc cccacccaat aaacccctcc
acgcgccccc
//
Виктория Соколик: В настоящее время мы с Prosolver продолжаем совершенствовать интерфейс программы TSP (с моим алгоритмом ![]() ), поэтому хочу с ней (программой) присоединиться к Вашей акции по продвижению в народ наших интеллектуальных разработок. Это может быть две сходные программы (Кушелева и Соколик) с немного разными в деталях алгоритмами, интерфейсами и возможностями, но базирующиеся на принципиально новом приёме создания моделей белка путём декодирования его структурного шаблона.
), поэтому хочу с ней (программой) присоединиться к Вашей акции по продвижению в народ наших интеллектуальных разработок. Это может быть две сходные программы (Кушелева и Соколик) с немного разными в деталях алгоритмами, интерфейсами и возможностями, но базирующиеся на принципиально новом приёме создания моделей белка путём декодирования его структурного шаблона.
Например, такой огромный белок как дистрофин (мутации в гене которого приводят к фатальным миопатиям) изучен в настоящее время только на
14%.
В моей программе TSP путём декодирования известной нуклеотидной последовательности (M18533 mRNA. Translation: AAA53189.1.) можно смоделировать структурный шаблон всего белка (3D) с четырьмя структурными доменами (их наличие установлено экспериментально):
...
Кушелев: Упростив аминокислоту до одной точки, наконец, удалось построить модель из 3685 элементов целиком ![]()
-- Nanoworld Laboratory. Alexander Kushelev. Peptide pikotechnology
-- http://nanoworld.narod.ru/
p = #()
angx = #(-24,180,60,-45,-45); angy = #(-10.89167,83,0,15,15); angz = #(92,120,60,110,110)
dx = #(2,1.5,1.6,2,2); dy = #(0.6,1,0.8,1,0.6); dz = #(-0.45,0,-0.6,-0.15,-0.45)
-- P11532
kk = #( 1,3,1,1,3,3,2,1,1,3,3,3,3,3,3,3,3,1,3,2,1,2,3,1,2,3,2,3,3,3,1,3,4,1,1,3,3,1,1,1,1,3,1,2,1,3,4,1,1,1,2,1,1,1,3,1,4,2
,4,3,3,4,2,3,3,3,2,1,2,3,3,3,1,4,1,3,1,1,1,2,4,4,3,1,1,1,3,3,3,3,3,4,3,3,2,3,3,1,1,2,3,2,3,3,3,4,3,3,3,1,3,1,3,2,1,1
,1,1,1,1,3,3,2,1,3,3,1,1,3,2,1,3,3,1,1,3,3,1,3,1,4,1,1,1,2,3,2,3,3,3,3,2,1,3,3,2,1,1,1,1,1,1,1,3,3,1,4,3,1,3,3,1,1,3
,3,3,1,2,1,2,3,1,1,3,3,4,3,1,1,1,2,1,2,3,2,4,3,3,2,1,1,1,1,3,3,3,3,1,2,1,3,2,1,3,3,3,3,3,3,1,1,3,2,3,1,1,1,1,3,1,1,1
,2,2,1,1,3,3,1,3,3,3,4,1,3,3,1,1,1,3,4,3,1,1,2,1,2,3,3,4,3,3,3,3,3,3,1,3,3,3,3,1,1,3,3,3,1,1,4,1,3,2,2,1,2,3,1,3,3,3
,1,3,1,3,2,1,1,1,3,1,1,2,1,3,3,3,1,1,1,3,1,3,2,4,1,2,3,3,2,1,3,1,3,3,3,3,1,1,2,3,1,3,2,1,1,1,3,3,2,1,4,1,3,3,3,2,3,3
,3,3,2,3,4,1,3,3,3,3,1,1,2,1,3,2,3,2,1,3,3,3,3,4,3,4,4,3,1,1,3,3,3,3,1,4,1,1,1,3,1,2,1,3,1,1,4,3,3,3,3,2,3,1,2,3,1,4
,3,2,2,2,3,3,2,3,3,3,3,3,3,2,3,1,1,1,3,1,2,3,2,3,1,3,1,1,1,2,3,1,1,3,3,3,1,3,3,3,3,3,3,1,3,1,1,3,1,3,4,3,1,1,3,1,1,2
,2,3,2,3,3,3,2,1,3,1,1,3,1,3,3,2,3,3,3,3,1,2,3,1,3,2,3,3,3,1,4,3,3,3,3,2,3,3,3,3,1,1,1,3,3,1,3,1,1,4,4,2,3,3,3,3,3,2
,3,1,2,3,3,3,1,3,3,3,3,1,2,1,2,3,2,1,2,1,1,3,3,1,2,3,1,1,1,3,3,3,3,1,1,3,1,3,1,3,3,3,3,3,3,1,1,3,3,3,2,1,3,2,3,3,3,3
,2,4,1,1,3,1,2,3,1,3,3,3,3,3,3,1,3,2,3,3,3,3,4,1,3,3,3,4,3,2,3,1,3,1,3,1,1,1,3,4,3,2,1,3,3,3,3,3,2,2,4,1,3,1,2,4,1,1
,1,4,3,2,1,4,3,1,3,1,4,3,1,3,3,3,1,3,3,3,3,1,3,2,2,1,3,2,1,3,1,1,1,3,1,2,2,2,2,1,2,3,2,1,3,2,2,3,4,4,1,2,1,3,1,1,4,2
,1,3,3,3,1,3,3,2,2,2,3,1,3,1,1,1,1,3,3,4,3,3,3,3,1,3,1,1,3,3,3,2,3,3,3,1,1,1,3,3,1,2,3,3,4,1,1,3,3,3,3,2,1,3,4,1,3,1
,1,1,2,1,3,3,3,3,1,3,1,2,1,2,3,3,3,1,1,1,3,3,4,3,3,1,1,3,2,3,1,1,4,4,3,1,1,4,3,1,3,3,3,2,3,1,1,3,3,1,2,3,3,4,1,1,4,1
,1,3,1,1,1,1,2,3,1,3,3,1,1,4,1,3,1,1,1,1,1,3,1,3,3,1,2,3,3,1,1,1,1,2,3,3,3,3,1,1,1,3,1,3,1,1,1,2,2,1,2,2,2,3,3,3,1,3
,3,3,3,1,3,3,1,1,4,2,2,3,3,3,3,3,3,3,2,3,3,3,3,1,2,1,4,3,1,3,2,3,2,1,1,1,4,3,2,1,3,4,1,3,2,3,3,3,1,3,1,3,3,3,4,1,1,3
,1,3,1,2,1,2,3,3,1,3,1,2,2,1,1,3,1,1,1,1,3,1,1,1,2,1,1,1,1,2,3,1,3,1,1,2,3,3,3,3,1,1,1,3,3,1,1,1,1,3,1,4,3,1,1,3,3,3
,3,3,4,3,1,3,3,3,1,2,1,3,1,1,1,3,4,3,1,1,4,1,3,4,1,3,3,3,1,4,3,3,3,2,3,3,3,3,3,1,2,1,1,1,1,1,1,1,1,4,3,1,3,3,3,1,2,1
,1,3,1,3,3,1,2,3,3,1,3,1,2,3,1,4,1,3,1,1,3,3,3,3,3,3,4,1,1,3,1,3,1,3,4,3,2,3,3,2,3,1,1,4,3,1,1,3,3,3,1,3,3,3,1,2,3,1
,1,3,2,1,3,1,3,3,3,4,1,1,2,1,3,3,2,1,2,1,3,3,4,3,3,1,2,3,1,3,3,3,1,3,1,1,3,1,1,1,3,1,1,3,1,3,1,1,1,1,1,2,3,1,1,3,3,2
,1,1,1,3,3,2,2,1,1,1,3,1,1,2,3,3,3,3,1,3,3,1,3,3,3,3,3,3,3,2,3,3,3,1,3,2,3,3,1,1,1,3,3,3,3,1,1,3,3,3,3,4,3,4,3,1,3,3
,1,3,2,3,3,1,2,3,3,3,2,3,2,2,3,1,1,3,3,1,3,3,3,3,2,1,1,1,1,1,1,1,1,3,1,4,3,4,3,1,1,3,1,3,3,3,1,2,3,1,3,1,3,1,2,1,1,1
,3,2,1,1,1,2,3,3,2,3,3,3,3,3,1,3,3,1,3,3,1,2,3,1,3,1,3,1,4,2,3,2,3,3,3,1,1,2,3,2,1,3,1,2,3,1,3,1,2,1,2,1,1,2,2,3,1,2
,1,1,3,1,2,1,3,1,3,3,1,2,3,3,3,3,1,1,3,2,3,3,1,3,2,1,1,3,1,1,3,3,1,1,1,1,3,1,1,1,3,3,3,1,3,1,3,1,1,1,1,1,2,1,3,1,1,1
,1,2,3,3,3,2,1,1,4,1,2,3,3,1,3,1,3,1,1,3,1,3,3,3,1,2,3,3,1,1,3,3,3,3,1,1,3,3,3,1,4,1,1,3,1,3,3,1,4,3,1,3,3,3,2,1,3,3
,3,3,3,1,1,1,1,3,2,3,1,1,3,2,1,3,3,1,4,3,2,3,3,3,1,1,3,3,3,3,4,1,1,1,1,3,2,1,3,2,1,3,4,3,1,3,2,2,1,2,1,2,3,3,3,4,1,1
,3,3,3,4,3,3,4,1,3,3,4,3,1,4,2,1,3,2,3,1,3,2,1,3,1,1,4,3,3,1,3,3,3,3,3,3,2,2,3,1,3,1,3,3,3,1,4,2,2,1,2,2,3,3,1,3,1,1
,1,3,1,1,3,1,1,3,1,1,2,1,3,1,3,1,1,2,3,1,4,2,3,2,3,1,3,1,2,1,3,2,2,3,3,2,1,3,3,3,1,3,3,3,3,1,1,2,1,3,3,3,3,1,3,1,3,1
,1,4,1,4,1,3,1,2,1,2,2,1,1,1,3,2,3,1,1,1,1,1,4,1,4,3,3,3,1,3,3,4,3,3,1,1,2,3,1,1,1,2,2,3,1,1,3,3,3,1,1,3,1,1,3,1,1,3
,3,3,1,1,3,4,1,1,1,2,1,1,1,3,1,3,1,2,3,1,3,3,2,1,3,1,3,1,1,3,3,3,1,4,3,1,3,3,1,2,3,4,3,1,2,1,2,1,4,1,3,2,3,1,3,2,2,1
,1,1,2,1,1,3,1,1,1,1,3,3,2,1,1,3,1,2,1,1,1,3,2,3,2,1,3,2,1,3,3,1,3,2,1,1,1,3,3,1,1,3,1,1,1,3,1,2,3,2,3,3,1,3,3,2,4,1
,3,3,3,1,1,4,4,3,4,3,1,3,1,1,3,3,3,1,3,3,1,3,1,3,3,2,3,3,1,1,3,3,2,1,1,3,3,3,3,1,2,3,1,3,1,3,1,3,2,1,2,3,1,1,4,3,1,2
,3,3,3,3,1,1,3,1,1,3,3,3,3,3,1,3,2,3,3,3,3,1,1,3,1,1,1,1,1,3,3,3,1,4,3,1,1,3,1,3,3,3,3,3,1,1,2,3,1,1,3,3,3,1,3,2,1,3
,3,3,4,3,1,1,1,1,3,1,1,4,3,2,4,3,1,3,3,1,1,1,3,1,3,4,1,2,1,2,4,1,2,3,1,1,1,1,1,1,1,1,4,3,3,1,1,3,3,3,1,3,2,3,1,1,3,2
,3,3,1,3,1,3,3,3,4,1,1,4,1,3,3,1,1,3,1,3,3,3,3,4,3,3,3,3,1,3,3,1,3,3,1,2,3,1,2,3,3,4,3,3,3,1,3,3,3,1,1,3,3,1,1,3,3,3
,1,3,1,3,1,1,3,4,1,3,2,3,3,3,2,3,3,1,2,3,4,3,1,3,3,3,1,1,1,2,2,2,1,3,3,2,4,3,4,3,1,4,1,2,1,3,3,1,1,1,3,3,1,3,1,3,3,3
,1,3,1,1,1,1,2,3,4,2,3,1,3,3,3,1,3,1,4,3,3,3,3,3,2,1,2,3,3,1,1,2,2,3,3,3,1,3,1,3,1,2,3,3,3,1,3,1,3,3,3,3,1,3,1,3,3,1
,3,1,1,3,1,3,4,1,4,3,3,3,1,3,2,1,3,2,3,4,3,3,2,3,1,3,1,2,3,2,3,1,3,3,2,1,3,3,1,2,1,4,3,4,4,1,1,1,1,1,3,1,4,2,1,3,3,1
,1,2,3,3,3,1,3,1,1,2,3,3,3,3,3,3,3,3,3,3,3,1,1,1,3,2,3,1,3,3,3,1,2,3,3,3,2,3,1,1,3,2,3,3,1,3,1,3,1,1,3,4,4,3,1,1,1,4
,1,1,2,3,1,3,3,3,3,3,3,2,2,1,4,3,2,3,3,1,2,1,2,3,1,3,3,3,3,3,3,1,1,1,1,2,3,1,1,1,2,1,3,1,3,3,3,3,1,3,3,2,3,3,3,3,3,2
,3,1,3,4,1,3,3,3,1,3,3,1,3,3,1,1,3,3,3,4,4,4,1,3,3,3,3,1,3,1,1,3,3,3,1,3,2,1,3,3,2,2,3,1,3,1,3,3,3,2,2,3,3,3,3,3,4,3
,4,3,1,3,1,3,3,4,1,3,1,1,1,3,3,2,1,3,1,2,4,1,1,1,3,3,3,4,1,3,1,1,1,4,2,1,3,3,3,3,1,4,1,2,1,1,3,1,2,3,3,2,4,1,3,3,2,1
,3,3,3,1,3,3,3,4,4,2,3,3,4,3,3,1,3,3,1,1,1,3,2,3,1,2,3,1,1,1,1,1,2,3,3,4,2,3,1,1,4,3,1,2,3,3,1,1,1,3,3,4,3,3,3,3,2,3
,2,1,1,4,1,4,3,1,3,1,3,1,1,1,1,1,1,1,1,1,2,2,1,1,3,1,3,1,1,3,1,1,1,3,3,1,3,1,3,1,3,3,1,3,1,1,1,1,3,3,1,3,3,2,1,3,4,3
,2,3,3,3,3,1,3,1,1,3,3,2,3,3,1,3,1,1,4,1,3,1,1,3,3,1,3,1,3,2,2,3,1,4,3,3,1,3,3,3,1,1,1,3,2,1,1,3,1,1,3,1,2,1,1,1,3,1
,3,2,2,3,2,1,3,1,3,1,2,3,1,1,1,1,1,3,1,1,1,1,1,1,2,3,2,3,4,2,3,1,1,1,4,3,3,1,4,3,3,3,2,3,3,1,3,3,1,1,1,2,2,1,3,1,3,1
,3,1,3,1,3,3,3,1,4,3,1,2,1,3,3,1,3,3,3,3,3,3,4,3,1,1,1,4,1,4,3,1,3,3,1,1,3,2,3,3,3,2,3,1,3,1,2,1,3,3,1,3,1,3,1,1,2,3
,1,1,1,2,2,3,1,4,1,3,3,1,3,1,1,3,3,3,3,3,3,1,2,3,3,3,1,1,4,3,3,1,1,3,3,1,4,3,1,4,3,3,1,3,3,2,1,4,3,3,3,3,1,3,1,1,1,1
,1,1,3,3,3,4,3,1,3,1,1,3,1,1,3,1,3,1,3,3,1,1,1,1,3,4,1,3,3,4,1,3,3,4,4,1,2,1,4,3,3,3,3,3,1,4,1,2,3,3,2,1,1,3,2,2,3,1
,1,1,1,3,2,3,1,1,1,1,1,3,1,3,3,3,3,3,2,1,1,3,3,3,1,3,2,2,2,3,4,2,1,1,3,1,3,2,2,1,3,1,1,1,1,1,3,1,4,3,3,1,1,3,1,1,3,1
,3,4,3,2,2,1,1,3,1,1,1,3,3,1,1,3,3,1,1,4,1,1,3,1,1,1,3,3,2,3,1,1,3,3,3,1,1,3,3,3,1,1,4,3,1,4,1,1,1,4,1,3,3,1,4,1,1,2
,1,1,1,1,4,1,3,1,1,3,1,3,1,3,3,1,1,1,3,1,1,2,3,2,2,3,3,4,3,4,3,1,1,4,1,1,1,3,1,3,3,1,1,3,1,3,1,1,3,1,1,2,4,3,1,1,1,3
,4,3,1,4,1,1,3,1,1,3,4,1,4,1,1,1,1,2,1,1,1,4,3,3,1,1,1,1,3,3,2,2,3,1,1,3,3,1,4,3,1,1,3,1,1,1,3,1,1,4,2,1,3,4,1,1,3,1
,1,1,1,3,3,2,3,1,1,1,3,1,3,1,2,1,1,1,1,3,3,3,1,4,3,3,1,3,1,2,3,3,1,3,1,1,3,1,2,3,4,1,1,1,3,1,1,3,1,1,1,4,2,3,2,3,3,1
,1,1,1,1,1,1,1,3,3,1,1,1,1,3,1,4,1,3,3,3,3,1,1,3,3,3,1,1,4,1,3,1,1,1,3,1,1,1,1,3,1,1,4,3,1,3,4,1,1,4,4,3,3,3,3,4,2,2
,2,4,1,1,3,1,4,3,3,3,3,1,1,3,1,4,3,3,2,3,1,3,1,1,1,3,1,3,1,1,3,4,2,3,2,2,2,3,3,1,1,1,1,4,1,1,3,4,3,3,3,1,3,3,2,3,1,1
,3,3,3,2,1,3,4,1,3,1,3,1,2,3,1,4,1,1,1,3,3,3,3,3,1,2,1,1,3,4,1,1,1,2,1,1,1,3,4,3,1,1,1,1,4,1,4,1,1,4,1,3,4,3,3,2,3,3
,1,1,3,1,1,3,3,1,1,1,3,1,3,2,1,3,2,1,1,1,1,3,2,1,1,3,3,3,1,1,1,3,1,1,3,3,3,3,2,3,2,3,1,3,3,1,1,3,1,1,4,3,3,1,3,4,3,2
,2,2,3,3,3,2,1,3,1,1,2,2,3,1,3,3,2,1,3,1,3,3,4,1,3,1,2,1,1,1,4,2,4,1,3,1,3,1,4,1,1,1,3,3,1,3,3,4,1,1,1,1,2,2,3,3,4,3
,1,4,1,3,1,3,2,1,3,3,3,3,2,1,3,3,3,3,3,1,1,2,2,3,1,3,1,1,3,2,3,3,2,3,3,1,1,3,3,3,1,1,2,3,3,3,3,1,3,1,1,3,1,1,3,3,1,1
,1,1,1,1,4,1,1,3,3,3,3,1,1,1,1,3,1,3,1,3,1,3,3,4,1,2,1,3,1,2,2,3,3,1,3,3,1,1,3,4,3,2,3,3,1,3,2,1,1,1,1,3,3,3,1,4,1,2
,4,4,1,3,3,3,1,1,1,1,3,1,1,3,1,4,3,3,1,1,3,3,1,1,1,2,4,3,3,1,3,1,1,4,3,1,1,1,3,1,4,3,1,1,3,3,1,4,1,2,1,3,1,1,2,1,1,4
,4,1,3,1,1,2,1,1,3,4,3,1,2,4,4,1,3,3,3,1,3,2,1,1,1,1,1,3,1,3,1,4,1,2,4,3,1,3,3,3,4,1,1,1,3,1,3,3,3,1,3,3,1,1,1,2,1,2
,4,3,1,1,4,1,1,3,1,1,1,1,1,3,3,2,3,2,3,3,1,3,2,1,2,1,3,1,1,2,1)
peptide = mesh vertices: #([0,0,0]) \
faces: #() \
wirecolor: [255,255,255]
element0 = copy peptide
for k = 1 to 3685 do(
element = copy element0
attach peptide element;
peptide.pivot = [0,0,0]; move peptide [dx[kk[k]],dy[kk[k]],dz[kk[k]]]
rotate peptide angx[kk[k]] [1,0,0]; rotate peptide angy[kk[k]] [0,1,0]; rotate peptide angz[kk[k]] [0,0,1])

Проверяем пикотехнологию на форуме Molbiol.ru
Flyamer,05.01.2012 23:40 пишет:Кушелев: Для этого нужна нуклеотидная кодирующая последовательность этого белка. Либо в стандарте Генбанка, т.е. файл с расширением dne, либо в стандарте fasta. Если дадите ссылку или сам файл, то я попробую с помощью этого скрипта: http://nanoworld.narod.ru/EMBLReader023L_20101113.txt
получить третичную структуру белка. Что касается сборки, то не гарантирую, но попытаюсь.Для скрипта достаточно этих строк:
CDS 145..825
ccgactcgga gcccctcggc ggcgcccggc ccaggacccg cctaggagcg caggagcccc
agcgcagaga
ccccaacgcc gagacccccg ccccggcccc gccgcgcttc ctcccgacgc
agagcaaacc gcccagagta gaagatggat tggggcacgc tgcagacgat cctggggggt
gtgaacaaac actccaccag cattggaaag atctggctca ccatcctctt catttttcgc
attatgatcc tcgttgtggc tgcaaaggag gtgtggggag atgagcaggc cgactttgtc
tgcaacaccc tgcagccagg ctgcaagaac gtgtgctacg atcactactt ccccatctcc
cacatccggc tatgggccct gcagctgatc ttcgtgtcca cgccagcgct cctagtggcc
atgcacgtgg cctaccggag acatgagaag aagaggaagt tcatcaaggg ggagataaag
agtggattta aggacatcga ggagatcaaa acccagaagg tccgcatcga aggctccctg
tggtggacct acacaagcag catcttcttc cgggtcatct tcgaagccgc cttcatgtac
gtcttctatg tcatgtacga cggcttctcc atgcagcggc tggtgaagtg caacgcctgg
ccttgtccca acactgtgga ctgctttgtg tcccggccca cggagaagac tgtcttcaca
gtgttcatga ttgcagtgtc tggaatttgc atcctgctga atgtcactga attgtgttat
ttgctaatta gatattgttc tgggaagtca aaaaagccag tttaacgcat tgcccagttg
ttagattaag aaatagacag catgagaggg atgaggcaac ccgtgctcag ctgtcaaggc
tcagtcgcta gcatttccca acacaaagat tctgacctta aatgcaacca tttgaaaccc
ctgtaggcct caggtgaaac tccagatgcc acaatggagc tctgctcccc
taaagcctca
aaacaaaggc ctaattctat gcctgtctta attttctttc acttaagtta gttccactga
gaccccaggc tgttaggggt
tattggtgta aggtactttc atattttaaa cagaggatat
cggcatttgt ttctttctct gaggacaaga gaaaaaagcc aggttccaca gaggacacag
agaaggtttg ggtgtcctcc tggggttctt tttgccaact ttccccacgt taaaggtgaa
cattggttct ttcatttgct ttggaagttt taatctctaa cagtggacaa agttaccagt
gccttaaact ctgttacact ttttggaagt gaaaactttg tagtatgata ggttattttg
atgtaaagat gttctggata ccattatatg ttccccctgt ttcagaggct cagattgtaa
tatgtaaatg gtatgtcatt cgctactatg atttaatttg aaatatggtc ttttggttat
gaatactttg cagcacagct gagaggctgt ctgttgtatt cattgtggtc atagcaccta
acaacattgt agcctcaatc gagtgagaca gactagaagt tcctagtgat ggcttatgat
agcaaatggc ctcatgtcaa atatttagat gtaattttgt gtaagaaata cagactggat
gtaccaccaa ctactacctg taatgacagg cctgtccaac acatctccct tttccatgac
tgtggtagcc agcatcggaa agaacgctga tttaaagagg tcgcttggga attttattga
cacagtacca tttaatgggg aggacaaaat ggggcagggg agggagaagt ttctgtcgtt
aaaaacagat ttggaaagac tggactctaa attctgttga ttaaagatga gctttgtcta
cttcaaaagt taaaaaaaaa aaaaaaaaaa aaaaaaaaa
//
Композиционный код, полученный по программе Prosolver:
1,3,1,1,4,4,1,4,1,4,4,3,4,1,3,1,1,1,1,3,2,1,1,1,1,1,1,1,1,3
,3,1,3,1,1,1,3,4,3,2,1,1,4,1,2,3,1,1,1,1,3,1,1,1,1,4,1,2,1,1
,1,1,4,1,1,3,1,1,1,1,1,1,1,1,4,2,1,1,4,1,4,1,1,4,1,4,2,4,1,2
,4,1,1,1,4,1,1,4,3,3,1,1,1,1,1,1,1,1,4,1,2,1,3,2,3,1,1,1,1,1
,1,3,1,1,1,1,1,1,3,1,1,4,1,1,1,1,2,1,1,1,1,1,4,1,1,1,3,1,1,1
,1,1,1,1,3,1,1,1,1,1,1,1,1,1,4,4,4,1,1,1,1,1,3,3,1,1,3,4,1,1
,3,4,1,4,1,4,1,1,3,1,1,2,4,1,1,3,2,4,3,2,3,1,1,4,4,3,1,3,3,1
,3,3,1,2,3,3,3,3,3,4,1,2,3,1,2,3
Скрипт для построения третичной структуры белка Cx26:
-- Nanoworld Laboratory. Alexander Kushelev. Peptide pikotechnology
-- http://nanoworld.narod.ru/
p = #()
angx = #(-24,180,60,-45,-45); angy = #(-10.89167,83,0,15,15); angz = #(92,120,60,110,110)
dx = #(2,1.5,1.6,2,2); dy = #(0.6,1,0.8,1,0.6); dz = #(-0.45,0,-0.6,-0.15,-0.45)
-- Cx26
kk = #(1,3,1,1,4,4,1,4,1,4,4,3,4,1,3,1,1,1,1,3,2,1,1,1,1,1,1,1,1,3
,3,1,3,1,1,1,3,4,3,2,1,1,4,1,2,3,1,1,1,1,3,1,1,1,1,4,1,2,1,1
,1,1,4,1,1,3,1,1,1,1,1,1,1,1,4,2,1,1,4,1,4,1,1,4,1,4,2,4,1,2
,4,1,1,1,4,1,1,4,3,3,1,1,1,1,1,1,1,1,4,1,2,1,3,2,3,1,1,1,1,1
,1,3,1,1,1,1,1,1,3,1,1,4,1,1,1,1,2,1,1,1,1,1,4,1,1,1,3,1,1,1
,1,1,1,1,3,1,1,1,1,1,1,1,1,1,4,4,4,1,1,1,1,1,3,3,1,1,3,4,1,1
,3,4,1,4,1,4,1,1,3,1,1,2,4,1,1,3,2,4,3,2,3,1,1,4,4,3,1,3,3,1
,3,3,1,2,3,3,3,3,3,4,1,2,3,1,2,3)
peptide = box length:1 width:1 height:1 position:[0,0,-.5] wirecolor:[250,250,250]; Converttomesh peptide
for k = 1 to 226 do(
element = Hedra family:1 scalep:100 scaleq:100 scaler:100 radius:1 pos:[0,0,0] p:0.4; Converttomesh element
attach peptide element; peptide.pivot = [0,0,0]; move peptide [dx[kk[k]],dy[kk[k]],dz[kk[k]]]
rotate peptide angx[kk[k]] [1,0,0]; rotate peptide angy[kk[k]] [0,1,0]; rotate peptide angz[kk[k]] [0,0,1])
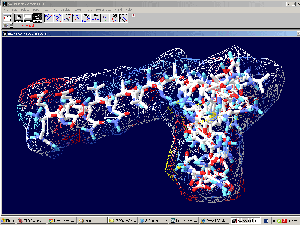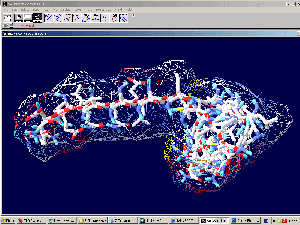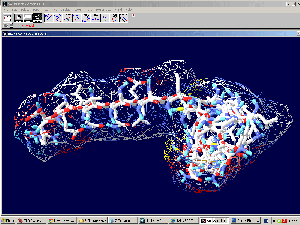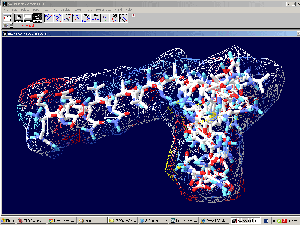
Третичная структура коннексина-26. В упрощённой пикотехнологической модели каждый аминокислотный остаток изображён одним 14-гранником.
Flyamer,06.01.2012 13:32 пишет:Кушелев: советую распечатывать субъединицы на 3D-принтере и пытаться составить из них четвертичную структуру. Конечно, можно попытаться сложить субъединицы в 3DS Max, как это удалось в случае спирали коллагена и слоя микротрубочки, образованной 13-ю субъединицами, состоящими из A,B и гамма-тубулина. Но в виртуальном пространстве сконструировать четвертичную структуру сложнее, чем из 3D-распечаток. 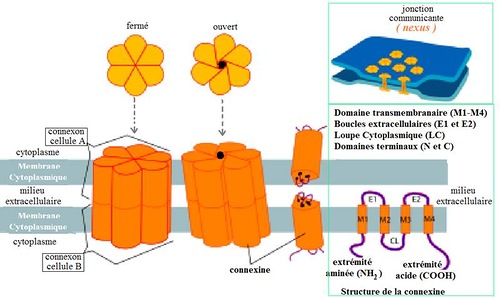
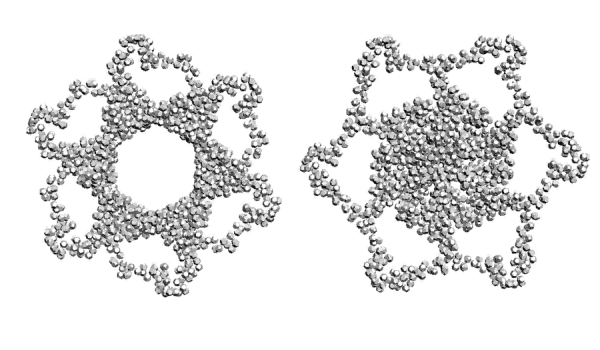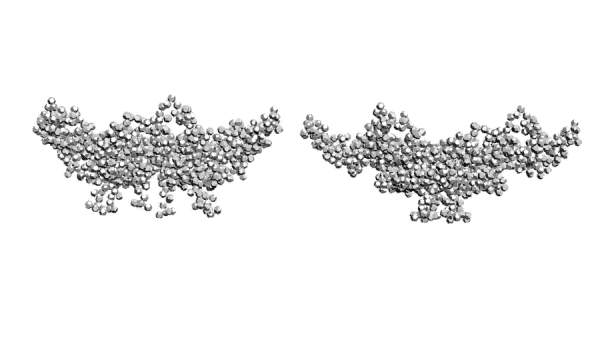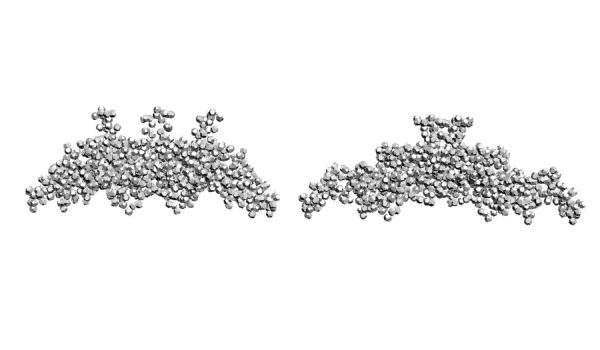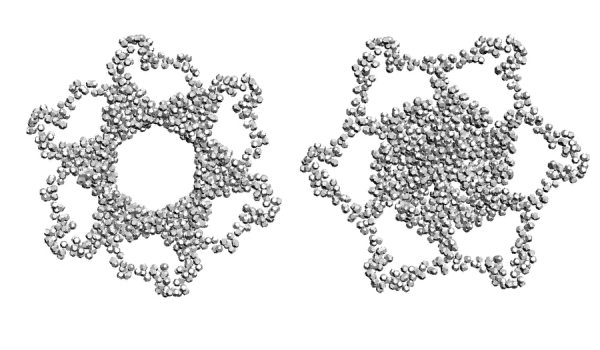
Flyamer пишет:
Кушелев: Давайте вместе посчитаем. Шаг альфа-спирали известен - 0,54 нм
Считаем число витков, и умножаем на 0.54 нм ![]()
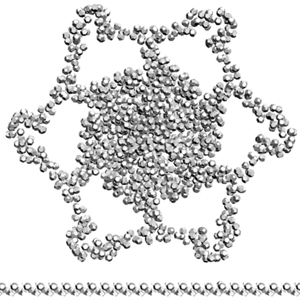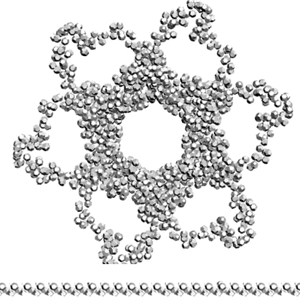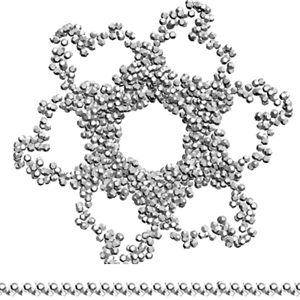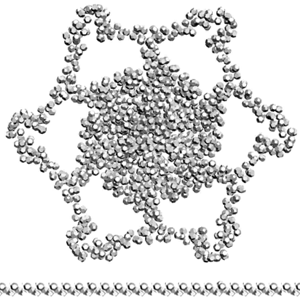

В этом ракурсе видно, что соседние коннексины должны входить в зацепление и двигаться до внешнего изгиба альфа-спирали соседнего коннексина. Четвертичную структуру удобнее собирать из 3D-распечаток субъединиц. В виртуальном пространстве сборку четвертичной структуры делать значительно сложнее. Хотя, если есть суперкомпьютер, специальные программы, шлем, перчатки, то может быть удобнее будет именно в виртуальном пространстве...
Flyamer,07.01.2012 17:03:
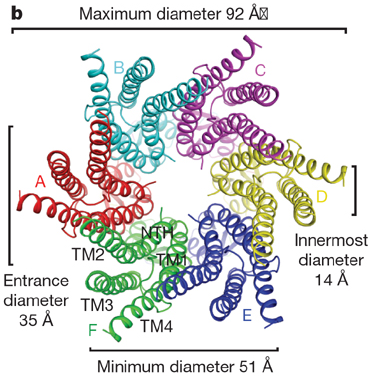
Кушелев:
Все так считают? ![]()

В случае зацепления коннексинов размер отверстия коннексона получается 1.5-2 нм, но нужно учитывать, что в этой модели не показаны радикалы аминокислотных остатков. А радикалы могут уменьшить диаметр отверстия на 0.5...1 нм. Так что имеет смысл построить менее упрощённую пикотехнологическую модель четвертичной структуры коннексона.
Flyamer,07.01.2012 17:38 пишет:
автор подгоняет результаты работы программы под реальность, все неточности "сбрасывает" на точность РСА. С этим работать невозможно.
Кушелев: Программа Пикотех не делает четвертичную структуру. Я же Вам объяснил, что четвертичную структуру приходится собирать вручную. При этом делать это в виртуальном пространстве 3DS Max без виртуальных перчаток, стереошлема и специальной программы очень неудобно. Поэтому с первого раза угадать четвертичную структуру коннексона не удалось. Да и со второго раза тоже, т.к. альфа-спирали налезают друг на друга. Похоже, что субъединицы должны быть несколько повёрнуты по другим осям.
Кстати, я не понял, что означает размер Entrance diameter 35A ? Это размер отверстия при максимальном увеличении диафрагмы-коннексона?
Тогда получается, что отверстие не может уменьшиться до нуля, как я пытаюсь изобразить в анимации? Тогда понятно, почему альфа-спирали налезают друг на друга. Они просто не могут до такой степени сблизиться, чтобы отверстие уменьшилось до нуля.
А с выводами Вы зря торопитесь. Модель ещё не доделана. Есть только пикотехнологическая модель третичной структуры, которая построена автоматически.
Flyamer,08.01.2012 13:01 пишет:
Кушелев: У меня тоже нет 3D-принтера, но я заказываю 3D-модели в специализированных мастерских. Это не очень дорого. Десяток субъединиц можно распечатать за 1000 рублей. Если интересно, то могу дать координаты мастерских 3D-печати.
Flyamer,08.01.2012 13:01 пишет:Откуда информация? Если в статье написано, что разрешение 3,5 ангстрема то что, они врут что ли?
Кушелев: Они не врут. Просто в статье может
быть указана рекордная точность для данной структуры, т.е. авторы спогли, например, определить расстояние между осями симметрии двух альфа-спиральных участков с точностью 1 анстрем. Вполне реально. Но длину этих же участков спирали они определили с точностью до витка, т.е. уже 5.4А, хвост белка РСА вообще не видит, а он может состоять из 30 аминокислотных остатков, т.е. кусок белка длиной ~100 ангстрем вообще не фиксируется методом РСА. Какой смысл повторять этот факт в каждой научной статье? Но у некоторых
читателей создаётся иллюзия, что точность РСА одинакова для всех частей молекулы белка. Так и рождаются мифы о невероятной точности РСА ![]()
Пока 97% белков не кристаллизуются. И, возможно, появится другой метод, не требующий кристаллизации, со сходным разрешением.
Кушелев: Этот метод (даже более точный) уже появился в 1992-ом году. Называется пикотехнология. Это не просто моделирование. Это - система модельных экспериментов. Именно из модельных экспериментов определяется форма молекулы белка, ведь программа Пикотех, складывает модель белка из моделей аминокислотных остатков, форма которых определена в модельном эксперименте...
Так что сравнение данных РСА и данных программы Пикотех в действительности является сравнением результатов разных экспериментов. При этом модели совпадают с точностью РСА, как менее точного из двух методов.
Мегалитические кольца Гамбии.

Цитата: Каждое кольцо состоит из 10-24 столбиков из латерита высотой 100—250 см. В общей сложности известно не менее 1000 таких каменных колец, им посвящён особый музей.
Хотя предназначение данных объектов вызывает споры, ЮНЕСКО посчитало возможным отнести их к памятникам Всемирного наследия (2006).
Координаты мегалитического комплекса в Синегамбии: 13.69,-15.522
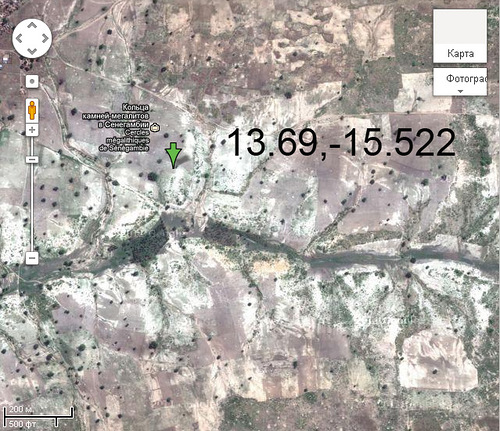
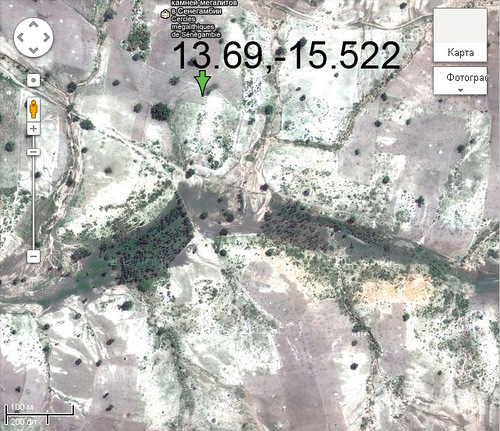

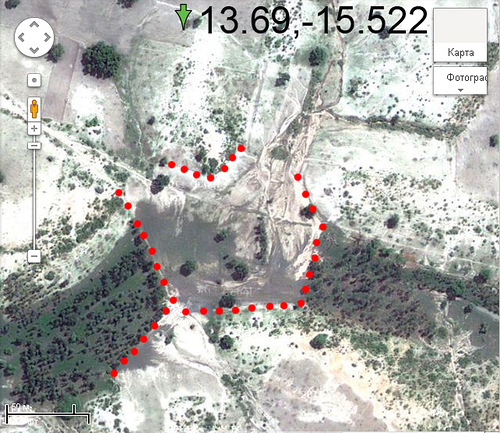
Здесь видно, что пересохшее русло реки подчищено, причём граница подчистки - ломаная линия...
Здесь уже видны разные признаки резонансной системы...

Каменная спираль на склоне...
Это в зоне Аркаима. Там три горы с каменными спиралями!
http://vymo.ru/arqaim/info:what
Каменные спирали могут быть крупными (десятки метров в диаметре)
Интересный диэлектрический генератор "трикветра"
Что бы это значило?
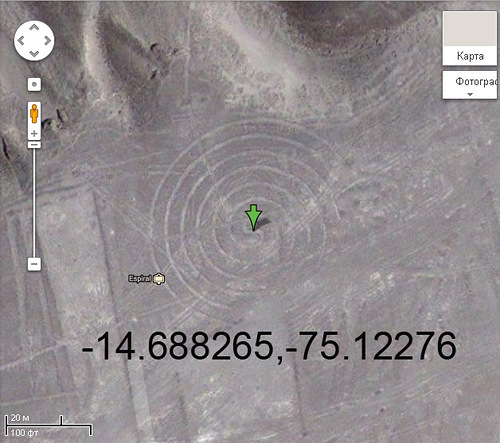
Каменная спираль на плато Наска. Диаметр около 50 метров. Судя по точности изготовления это вполне мог быть рабочий источник энергии выпаривательного комплекса...
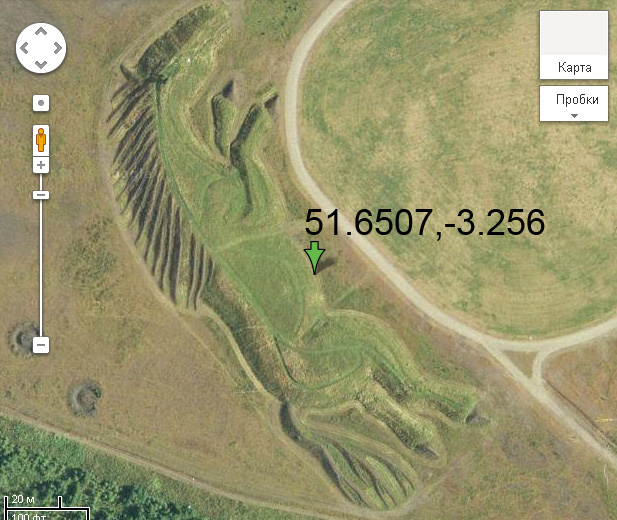
Координаты: 51.6507,-3.256
Любопытный конь-рельеф длиной более 100 метров. Интересно, сколько он весит и когда появился? В инете практически нет никакой информации об этом...

Пришельцы использовали искусственные тела?!

"Крепость" Святой Анны ... выпаривала исток Азовского моря!
Кушелев: Интересно. Нужно изучить...

Ого! Какая удача...
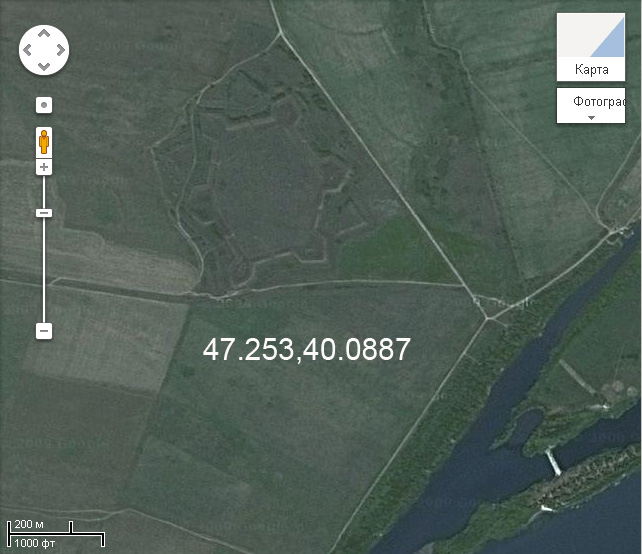
Координаты: 47.253,40.0887
Не нужно быть Шерлоком Холмсом, чтобы понять, что земляной вал построен не по этому кривенькому плану:
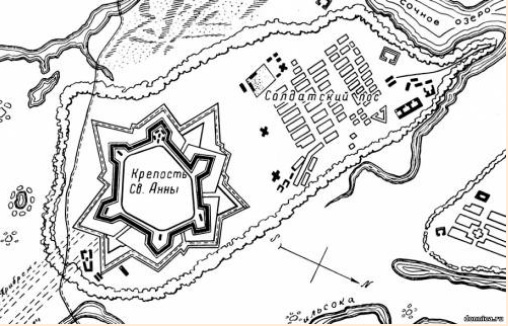
и не по этому:
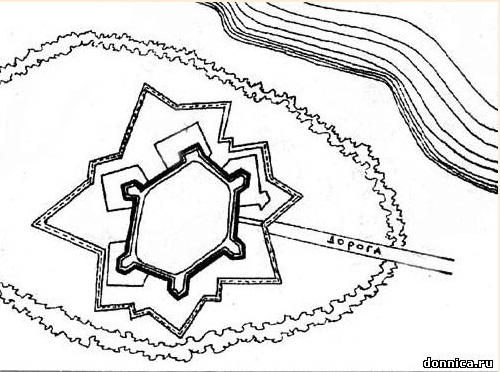
Люди вообще не насыпали земляной вал, а только реставрировали...
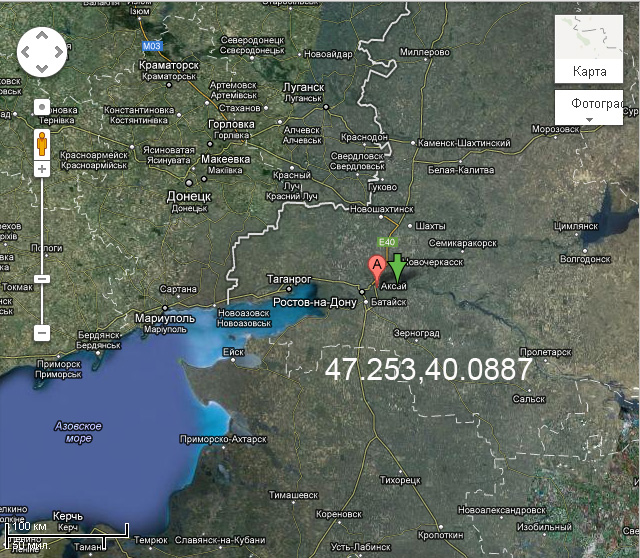
"Крепость" Святой Анны ... выпаривала исток Азовского моря ![]()
Приглашение к сотрудничеству
Лаборатория Наномир готова к любому взаимовыгодному сотрудничеству. У нас есть сторонники как явные, которые помогают морально и материально, есть очень много пассивных наблюдателей, есть и ярые противники, которые используют любые методы и средства (аморальные и просто преступные), чтобы уничтожить работу лаборатории и дискредитировать ее.
В одиночку внедрить технологии, выводящие цивилизацию на новый уровень, невозможно. Благодаря поддержке множества заинтересованных людей проделана огромная работа. Ознакомиться с её результатами можно изучив материал рассылки "Новости лаборатории Наномир". Люди науки могут изучить научные труды.
Вклад каждого не останется незамеченным в случае успеха в реализации научных проектов. Результаты совместной деятельности принадлежат участникам проекта пропорционально коэффициентам творческого и финансового участия.
В этом году были куплены рубиновые шарики для эксперимента на сумму ~1000 долл. В результате было сделано научное открытие, проверена защита диэлектрических резонаторов от перенапряжения. В этом же году, вероятно, можно будет создать микроволновую энергетику, т.к. удалось найти сырьё (рубин #8), из которого сделаны рубиновые шарики для эксперимента в Дубне.
28 сентября начался эксперимент по созданию "эликсира вечной молодости". Благодаря первому взносу (в размере 500 долларов) Золдракса и поддержке других соинвесторов. Продолжаются переговоры с потенциальными инвесторами по поводу финансирования этого проекта.
Созданы первые версии пикотехнологии, с помощью которой Александр Кушелев и Виктория Соколик сделали более10 научных открытий.
Сотрудничество может быть различным:
- участие в научных дискуссиях на форуме (конструктивное)
- совместное создание коммерческого продукта
- поиск инвесторов
- выступить менеджером по продаже готовых коммерческих продуктов
- конструктивные предложения по продвижению идей лаборатории Наномир
- содействие в проведении экспериментов и т.п.
- написание совместных научных статей и т.п.
- материальный вклад (денежный или обеспечение оборудованием и материалами)
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир.
+7-926-5101703 , +7-903-2003424 , +7-916-8265031 , Skype: Kushelev2009, mail: kushelev2011@yandex.ru
веб-мани: WM-кошелек R426964799301