
Выпуск 232
Лаборатория Наномир
Когда реальность открывает тайны,
уходят в тень и
меркнут чудеса ...
Три составные части успеха ноосферы

Изучить омолаживающий эффект модификации гормонального фона и частичной иммуносупрессии, которые развиваются при беременности, на модели гетерохронного парабиоза
ГУ «Институт геронтологии АМН Украины», Киев
Лаборатория
«Наномир», Москва
Старение организма – многофакторный процесс, обусловленный, в том числе, и утратой регенеративного потенциала стволовых клеток. Оказывается, существует прямая связь между химическими сигналами, присутствующими в старой крови, и утратой способности тканей к регенерации [ 1, 2, 3, 4 ]. Это, в большинстве случаев, сводит к нулю эффективность терапии с применением стволовых клеток в частности [ 5 ], и является одной из главных причин старения организма в целом. Следовательно, выявление и блокировка этих сигналов является важнейшей задачей антивозрастной терапии.
Угнетающее действие сыворотки крови старых людей и животных на рост клеток в культуре впервые было выявлено еще в 1921 году A. Carrel и A. H. Ebeling. Они показали, что рост культуры фибробластов цыплят зависит от возраста донора, сыворотку которого использовали для культуральной среды [ 6 ]. С тех пор с феноменом существования факторов старения, переносимых с кровью, сталкивалось большое количество исследователей. Так, например, японские авторы показали, что сыворотка крови старых людей способна подавлять пролиферацию Т-лимфоцитов in vitro. При использовании в качестве контролей сывороток пациентов с гепатомами, или сывороток беременных женщин, авторы установили, что факторы «старения», находящиеся в сыворотках старых людей, обладают уникальным механизмом супрессии пролиферации лимфоцитов, и, в отличие от других сывороток, способны неспецифически подавлять рост клеток на любом этапе активации [ 7 ]. Более того, в работе Бутенко Г. М. и др. было показано, что пациенты, сыворотка которых обладает выраженным ингибирующим действием в реакции бласттрансформации in vitro, имеют ограниченный срок жизни и умирают в течение ближайших 2-х лет с момента тестирования [ 8 ].
Подобный эффект был обнаружен на модели гетерохронного парабиоза ? в эксперименте у молодых мышей, партнеров по парабиозу, появляются выраженные возрастные изменения иммунологических функций, которые прогрессируют по мере сосуществования животных, и которые, очевидно, передаются с кровью [ 9 ].
В 2010 году израильскими учеными было показано, что беременность стимулирует регенераторные возможности организма матери. Установлено, что у беременных самок мышей возрастом 10-12 месяцев скорость восстановления печени после гепатэктомии увеличивается вдвое, в то время как у молодых мышей скорость регенерации печени не зависит от наличия беременности [ 10 ]. Авторы предполагают, что при беременности в крови появляются факторы, способные тормозить процессы старения.
Целью данного исследования является выявление фактора, появление которого в кровотоке беременных самок способствует улучшению процессов регенерации и тормозит возникновение возрастных нарушений иммунной системы.
Задачи проекта: на модели гетерохронного парабиоза, которая представляет собой химеру из двух животных разного возраста, которые имеют общую кровеносную систему, изучить влияние беременности на возникновение возрастных изменений иммунной системы у партнеров разного возраста.
Дизайн 1-го этапа эксперимента:
1. Животные ? мыши самки линии СВА/Са возрастом 3 мес (молодые), 10?12 мес (стареющие), 21?23 мес (старые).
2. Экспериментальная модель ? создание парабиотических пар.
3. Экспериментальные группы:
3.1. Проверка влияния беременности молодого партнера на процессы старения иммунной системы на модели гетерохронного парабиоза.
Молодые изохронные пары ? молодая мышь, сшитая с молодой ложнобеременной, 10 пар.
Молодые изохронные пары ? молодая мышь, сшитая с молодой беременной, 10 пар.
Гетерохронные пары ? молодая мышь, сшитая со старой, 10 пар.
Гетерохронные пары ? молодая беременная мышь, сшитая со старой, 10 пар.
3.2. Проверка влияния беременности стареющего партнера на процессы ускоренного старения иммунной системы у молодых партнеров по гетерохронному парабиозу.
Гетерохронные пары ? молодая мышь, сшитая со 10-12 мес ложнобеременной мышью, 10 пар.
Гетерохронные пары ? молодая мышь, сшитая со 10-12 мес беременной мышью, 10 пар.
Стареющие изохронные пары ? 10-12 мышь сшитая с 10-12 мес ложнобеременной мышью, 10 пар.
4. Схема проведения эксперимента:
4.1. Вазектомия самцов для обеспечения ложной беременности контрольных самок.
4.2. Проведение операций по созданию парабиотических пар.
4.3. Через 7 дней после проведения операций провести полноценное или ложное оплодотворение экспериментальных животных.
4.4. Через 15-20 дней после оплодотворения провести тестирование показателей состояния иммунной системы.
5. Тестируемые показатели:
5.1. Уровень прогестерона в плазме крови.
5.2. Визуальная проверка беременности (наличие плодов в матке, оценка предположительного срока).
5.3. Оценка состояния иммунной системы ? проверка массы тимуса, пролиферативной активности спленоцитов, субпопуляционного состава Т-клеток селезенки.
5.4. Сбор и криосохранение плазмы крови экспериментальных животных.
Дизайн 2-го этапа эксперимента:
При всём существующем на сегодняшний день наборе методик идентификация регуляторных факторов процессов старения является довольно трудной задачей. Известно достаточно большое количество случаев, когда попытки идентификации регуляторных факторов не давали никаких результатов. Для решения подобных задач нами разработан и апробирован набор оригинальных методик, позволяющий находить комбинации управляющих сигналов, что делает возможным обнаружение химической природы регуляторных факторов.
Поиск факторов старения сыворотки крови предполагает широкий скрининг с оценкой суммарной ингибирующей активности тестируемой сыворотки на культурах клеток in vitro; а также последующих за этим этапах разделения сывороточных фракций с использованием различных методологических подходов: ультрацентрифугирование, хроматография, высаливание, диализ, электрофорез, обработка ДНКазами, РНКазами, протеазами, специфическими антителами. С последующим тестированием на культурах клеток in vitro с целью выявления фракций, обладающих максимальным ингибирующим или стимулирующим эффектом.
Еще один возможный подход ? анализ факторов плаценты, обладающих эффектом «антистарения». Данный подход является более сложным для анализа, поскольку требует этапа разрушения клеток с последующим выделением биологически-активных фракций. Поскольку мы не знаем, к какому типу биологических молекул относятся активные факторы плаценты, достаточно сложно выбрать процедуру их экстракции. Поэтому, с нашей точки зрения, более логично начать с поиска факторов плазмы крови, которые появляются в крови беременных особей.
Выявление специфических факторов старения (или антистарения) организма позволит установить их структуру и разработать подходы к их нейтрализации в организме.
1. Анализ состава фракций плазмы крови, которые будут обладать ингибирующим или стимулирующим действием на рост и дифференцировку клеток in vitro. Разделение на плазмы на подфракции с использованием ультрацентрифугирования с использованием молекулярных фильтров. Обработка подфракций протеазами, ДНКазами, РНКазами, использование хроматографии, высаливания, диализа, электрофореза, и тестирование полученных подфракций на культурах клеток мыши in vitro. Основная цель фрагмента ? выявить ключевую молекулу(ы), обладающую(ие) искомой биологической активностью.
2. Анализ структуры искомой молекулы с использованием масс-спектрометрии или других соответствующих подходов в зависимости от предполагаемой природы полученной субстанции.
3. Проверка эффекта введения полученной субстанции in vivo на животных.
4. Разработка подходов к блокированию (элиминации) предполагаемой субстанции в организме.
Бюджет исследования.
|
|
Статьи расходов |
1 этап |
2 этап |
|
1. |
Зарплата |
4000 грн |
15000 грн |
|
2. |
Животные, корма, уход |
6000 грн |
4000 грн |
|
3. |
Проведение операций (наркоз, микрохирургический инструмент, постоперационный уход) |
6000 грн |
? |
|
4. |
Анализ иммунологических показателей экспериментальных животных (пластиковая посуда, среды, услуги проточного цитометра) |
4000 грн |
4000 грн |
|
5. |
Поддержание клеточных культур для анализа активности факторов сыворотки крови (культуральная посуда, среды, эмбриональная сыворотка, стерилизующие фильтры, культуральные добавки, и пр.) |
? |
20000 грн |
|
6. |
Фракционирование плазмы крови экспериментальных мышей (молекулярные ультрацентрифужные фильтры, расходные материалы для электрофореза, диализа, хроматографии, ферменты для установления природы искомой молекулы (ДНКазы, РНКазы, протеазы)). |
? |
20000 грн |
|
7. |
Анализ структуры искомой молекулы (услуги масс-спектрометра, секвенатора, аминокислотного анализатора) |
? |
10000 грн |
|
8. |
|
|
|
Литература:
1. Conboy I.M., M.J. Conboy, A.J. Wagers, E.R. Girma, I.L. Weissman, T.A. Rando. Rejuvenation of aged progenitor cells by exposure to a young systemic environment // Nature, Vol 433, 17 February 2005.
2. S.R. Mayack, J.L. Shadrach, F.S. Kim & A.J. Wagers. Systemic signals regulate ageing and rejuvenation of blood stem cell niches. Nature, Vol 463, 28 January 2010.
3. Hiroshi Kondo, Yumiko Yonezawa and Hideki Ito. Inhibitory Effects of Human Serum on Human Fetal Skin Fibroblast Migration: Migration-Inhibitory Activity and Substances in Serum, and Its Age-Related Changes In Vitro // Cellular & Developmental Biology. Animal Vol. 36, No. 4 (Apr., 2000), pp. 256-261.
4. Зуев В.А., Автандилов Г.Г., Игнатова Н.Г., Быковская С.И. Индуцированное старение мышей // Бюлл. Эксп. Биол. Мед. – 2003. – Т. 136, № 3. – С. 286-287.
6. Carre, A., and Ebeling, A. H., J. Heterogenic serum, age, and multiplication fibroblasts // Exp. Med., 1921, xxxiv, 599.
7. Masato Tsujimoto, Tadaaki Tsuda, Masahiro Ohata. Mechanisms of Inhibitory Effect of Serum from Old Subjects on Lymphocyte Responsiveness to Mitogen // Japanese journal of geriatrics.? Vol.18 , No.5(1981)pp.361-369
8. Бутенко Г. М., Зайченко А. П. Использование иммунологических показателей для прогнозирования течения старения и болезней // Геронтология и гериатрия: Ежегодник. 1990: Превентивная геронтология и гериатрия, К., 1991. – С. 67-71.
10. Y. Gielchinsky, N. Laufer, E. Weitman, et al. Pregnancy restores the regenerative capacity of the aged liver via activation of an mTORC1-controlled hyperplasia/hypertrophy switch // Genes Dev. 2010 24: 543-548

Приблизительно
так будет выглядеть рубиновая "вечная лампочка".
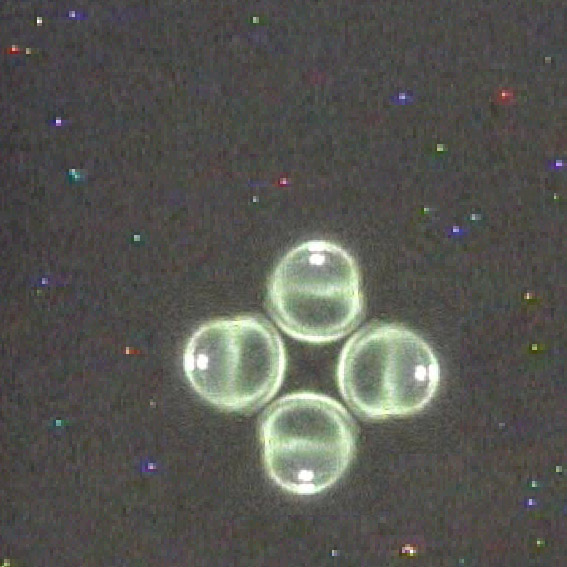
Это почти реальный кадр.

Но в реальности существует только один шарик из ИАГ-Cr, настроенный в резонанс с лучом из ускорителя в Дубне.
У меня есть только три этих видеокадра со второго эксперимента в Дубне. Второй эксперимент в конце мая был проверочным. Специалисты решили убедиться, что в гранатовом шарике N2 действительно происходит свечение на колебательной моде "шепчущей галереи". И им это удалось. К сожалению, работа ускорителя снова прервана на неопределённый срок. Если бы вместо одного гранатового шарика у специалистов был рубиновый генератор, который при комнатной температуре имел ту же резонансную частоту, то микроволновая энергетика, вероятно, уже была бы создана. Но для этого нужно иметь много рубиновых шариков. Купить их инвесторы не могут, т.к. "много шариков" - это дороже 5000 долл. Плавно менять температуру специалисты из Дубны не берутся. Объясняют это отсутствием технических возможностей. Аппаратура для точного дистанционного управления температурой и резонансной частотой рубинового генератора может стоить ещё дороже, чем сотня рубиновых шариков. Есть ли возможность удешевить проект? Да. В том случае, если удастся найти сырьё, из которого можно сделать сотни рубиновых шариков. В настоящее время мне неизвестно, где можно приобрести рубин N11 оптического качества, из которого можно делать шарики размером 9-12 мм.
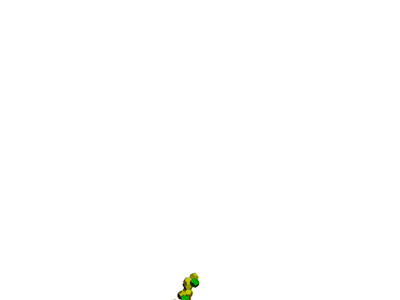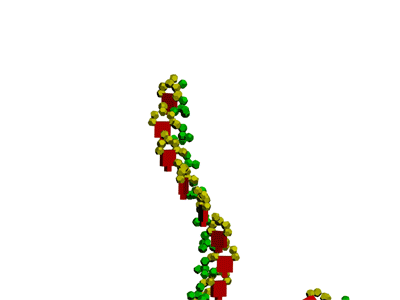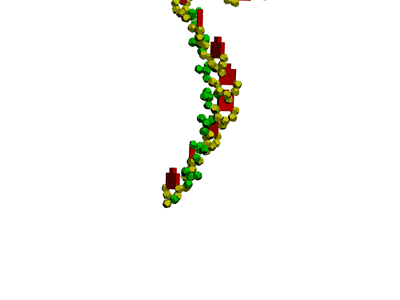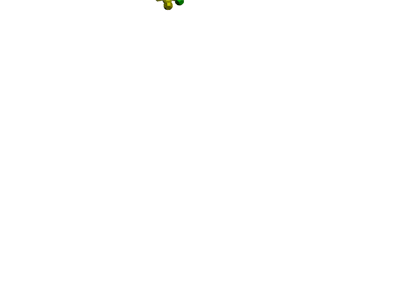
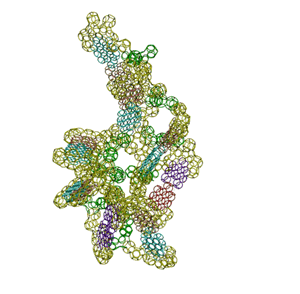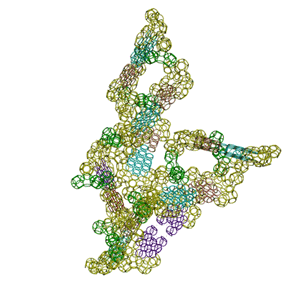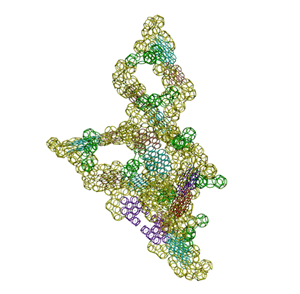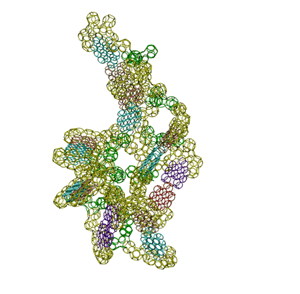
Модель тРНК ещё не доделана, но уже понятно, как она работает. Осталось уточнить нюансы...
Модель перекрестия тРНК без вариабельной петли (фрагмент, состоящий из 6 комплементарных пар нуклеотидов).
Повернуть
двойную спираль на 90 градусов можно поворотом на композиционный угол-2
(на 120 градусов). Второй поворот на 90 градусов тоже можно сделать на
одном нуклеотиде. При этом получится небольшое смещение спирали. Третий
поворот тоже можно осуществить на одном нуклеотдиде. Но последний
поворот потребует компенсировать смещение, поэтому на одном нуклеотиде
не удастся его осуществить...
Скрипт, который строит пикотехнологическую (кольцегранную) модель ДНК/РНК:
p = #(); p1 = #(); p2 = #(); aa = #(); ax = #(); ay = #(); r = #()
; nuclcol = #(); gp = #(); gn = #(); gg = #(); anuk = #()
-- adenosine, cytosine, guanine, timidine, uracil, inosine, pseudouracil
--, dihydrouracil, methyl inosine, x-circles maximum, w-methyl-2-guanine, v-methyl-1-guanine
nucl = #("a","c","g","t","u","i","p","d","m","x","w","v")
nuclcolor = #([200,0,0],[200,100,100],[0,200,200],[0,0,200],[100,0,200],[200,0,200]
,[150,0,0],[100,50,0],[0,80,50],[100,100,100],[0,200,0],[200,200,0],[255,255,255],[0,0,0])
--****************************************************
seq = #("a","c","c","a","c","c","u","g","c","u"
-- 11
,"c","a","g","g","c","c","u","u","a","g"
-- 21
,"c","p","t","g","g","c","c","u","c","d"
-- 31
,"g","g","a","g","a","g","g","g","p","m"
-- 41
,"c","g","i","u","u","c","c","c","u","c"
-- 51
,"w","c","g","c","g","a","d","g","g","c"
-- 61
,"d","g","a","u","g","c","g","v","u","g"
-- 71 76
,"u","g","c","g","g","g")
--****************************************************
complement = #("0","0","0","0","g","g","g","c","g","u"
-- 11
,"g","u","c","c","g","g","0","t","p","0"
-- 21
,"0","0","0","0","0","0","0","0","0","w"
-- 31
,"0","0","0","c","u","c","c","c","i","0"
-- 41
,"0","0","0","0","0","0","0","0","0","0"
-- 51
,"0","g","c","g","u","a","g","d","0","0"
-- 61
,"0","0","0","0","0","0","0","0","0","0"
-- 71 76
,"0","0","0","0","0","0")
--****************************************************
ci = #(0,0,0,0,1,1,1,1,1,1
-- 11
,1,1,1,1,1,1,0,2,3,0
-- 21
,0,0,0,0,0,0,0,0,0,0
-- 31
,0,0,0,1,1,1,1,1,3,0
-- 41
,0,0,0,0,0,0,0,0,0,0
-- 51
,0,1,1,1,4,5,6,6,0,0
-- 61
,0,0,0,0,0,0,0,0,0,0
-- 71 76
,0,0,0,0,0,0)
--****************************************************
anglecomp1 = #(0,0,0,0,0,0,0,0,0,0
-- 11
, 0, 0, 0, 0, 0, 0,
0, 120, 0, 0
-- 21
,-120,
0, 0, 0, 0,
0, 0, 0, 0, 0
-- 31
, 0, 0, 0, 0, 0, 0, 0, -20, -40, 40
-- 41
,
0, -30, 0, 0, 0,
0, 0, 0, 0, 0
-- 51
,
0, 0, 0, 0,
0, 0, 0, 0, 120, 0
-- 61
,
0, 0, 0, 0,
0, 0, 0, 0,
0, 0
-- 71 76
, 0, 0, 0, 0, 0, 0)
anglecomp2
= #(0,0,0,0,0,0,0,0,0,0
-- 11
, 120, 0, 0, 0, 0, 0, 0, 0, -30, -60
-- 21
,
120, 0, 0, 0, 0,
0, 0, 0, 0, 0
-- 31
,
0, 0, 0, 0,
0, 0, 0, 0, 0,-120
-- 41
, 0, 120,
0, 0, 0, 0, 0, 0, 0, 0
-- 51
,
0, 0, 0, 0,
0, 0, 0, 0, 60,
0
-- 61
,0 , 0, 0,
0, 0, 0, 0,
0, 0, 0
-- 71
76
, 0, 0, 0, 0, 0, 0)
--****************************************************
trnk = torus radius1:0.1 radius2:0.04 segs:3 sides:3 position: [0,0,0] wirecolor:[200,200,200]
Converttomesh trnk
--****************************************************
px
= #(4.2,-4.2,4.2,-4.2,-4.2,4.2,-4.2,4.2); py =
#(-4.2,-4.2,4.2,4.2,4.2,4.2,-4.2,-4.2); pz =
#(4.2,4.2,4.2,4.2,-4.2,-4.2,-4.2,-4.2)
pa = #(35.27,35.27,-35.27,-35.27,35.27,35.27,-35.27,-35.27); pb = #(45,-45,45,-45,45,-45,45,-45)
-- PH2
pindex = #(1,0,0,1,0,1,1,0, 0,0,0,0,0,0,0,0, 1,1,1,1,1,0,1,1, 0,1,1,1,1,1,1,1, 1,1,1,0,1,0,1,1, 1,0,1,1,0,1,1,1)
pindex2 = #(1,0,0,1,0,1,1,0, 0,0,0,0,0,0,0,0, 1,1,1,1,1,0,1,1, 0,1,1,1,1,1,1,1, 0,0,0,0,0,0,0,0, 0,0,0,0,0,0,0,0)
trnk = torus radius1:0.1 radius2:0.04 segs:3 sides:3 position: [0,0,0] wirecolor:[200,200,200]
Converttomesh
trnk
newmat = multimaterial name:"MyMultiMat" numsubs: (14)
newmat[1].faceted = on
newmat[2].faceted = on
newmat[3].faceted = on
newmat[4].faceted = on
newmat[5].faceted = on
newmat[6].faceted = on
newmat[7].faceted = on
newmat[8].faceted = on
newmat[9].faceted = on
newmat[10].faceted = on
newmat[11].faceted = on
newmat[12].faceted = on
newmat[13].faceted = on
newmat[14].faceted = on
newmat[1].diffuse = (color 200 0 0)
newmat[2].diffuse = (color 200 100 100)
newmat[3].diffuse = (color 0 200 200)
newmat[4].diffuse = (color 0 0 200)
newmat[5].diffuse = (color 100 0 200)
newmat[6].diffuse = (color 200 0 200)
newmat[7].diffuse = (color 150 0 0)
newmat[8].diffuse = (color 100 50 0)
newmat[9].diffuse = (color 0 80 50)
newmat[10].diffuse = (color 100 100 100)
newmat[11].diffuse = (color 0 200 0)
newmat[12].diffuse = (color 200 200 0)
newmat[13].diffuse = (color 255 255 255)
newmat[14].diffuse = (color 0 0 0)
trnk.material = newmat[10]
for k = 8 to 15 do(
for
ll = 1 to 8 do(if pindex[ll] > 0 then (p[ll] = torus radius1:4.8
radius2:0.4 segs:20 sides:12 position: [px[ll],py[ll],pz[ll]]
wirecolor:[0,200,0]
rotate p[ll] pa[ll] [1,0,0]; rotate p[ll] pb[ll] [0,1,0])); g0 = group #(p[01],p[04],p[06],p[07])
for
ll = 17 to 24 do(if pindex[ll] > 0 then (p[ll] = torus radius1:4.8
radius2:0.4 segs:20 sides:12 position: [px[ll-16],py[ll-16],pz[ll-16]]
wirecolor:[0,200,0]
rotate p[ll] pa[ll-16] [1,0,0]; rotate p[ll] pb[ll-16] [0,1,0])); g2 = group #(p[17],p[18],p[19],p[20],p[21],p[23],p[24])
rotate g2 60 [-1,-1,1]; move g2 [-8.4,-8.4,8.4]
for
ll = 25 to 32 do(if pindex[ll] > 0 then (p[ll] = torus radius1:4.8
radius2:0.4 segs:20 sides:12 position: [px[ll-24],py[ll-24],pz[ll-24]]
wirecolor:[0,200,0]
rotate p[ll] pa[ll-24] [1,0,0]; rotate p[ll] pb[ll-24] [0,1,0])); g3 = group #(p[26],p[27],p[28],p[29],p[30],p[31],p[32])
rotate g3 60 [-1,1,-1]; move g3 [-8.4,8.4,-8.4]; g6 = group #(g0,g2,g3); g6.pivot = [0,0,0]
rotate g6 -45 [0,0,1]; rotate g6 -15.5 [1,0,0]; move g6 [-37.5,-9.5,11.5]; ungroup g6; ungroup g3; ungroup g2; ungroup g0
select #(p[01],p[04],p[06],p[07],p[17],p[18],p[19],p[20],p[21],p[23],p[24],p[26],p[27],p[28],p[29],p[30],p[31],p[32])
macros.run
"Modifier stack" "convert_to_Mesh"; element2 = copy p[1] wirecolor: [0,200,0]
for ll = 2 to 32 do(if pindex2[ll] > 0 then (attach element2 p[ll])); delete p[1]
element2.material = newmat[11]
-- rybose
x
=
#(6,-6,0,0,0,0,15,-15,23.5,-23.5,25,-25,20,-20,19,-19,19,-19,22.5,-22.5,22.5,-22.5,10,-10,10,-10,15.5,-15.5,15.5,-15.5,0,6,-6,0,-5.5,5.5,0,-23.7,-16.5,-26
,-23.5,-18,-15.5,-26,-29.53,-31.51,-21.611,-23.59,-21.611,-19.631,-29.53,-27.55,-43.389,-43.39,-37.45,-37.45,-31.51,-31.51,-37.45,-37.45)
y
=
#(-58,-58,-58,-58,-49,-49,-55,-55,-47,-47,-37.5,-37.5,-26.5,-26.5,-50.5,-50.5,-50.5,-50.5,-32,-32,-32,-32,-47,-47,-47,-47,-38.5,-38.5,-38.5,-38.5,-55,-63
,-63,-69,-60.5,-60.5,-66,-55.7,-48,-58,-48,-58,-56,-50,-28.049,-26.864,-25.754,-24.57,-32.563,-33.747,-34.857,-36.042,-26.864,-19.981,-23.46,-16.576,-19.981
,-26.864,-23.385,-30.268)
z
=
#(0,0,6,-6,7,-7,0,0,0,0,0,0,0,0,6,6,-6,-6,6,6,-6,-6,7,7,-7,-7,7,7,-7,-7,-13.5,-8.5,-8.5,-10.5,-16,-16,-18,-17.5,-13,-10,-15.5,-8,-15.5,-8,17.242,9.165
,15.637,7.56,5.902,13.98,7.507,15.584,9.165,4.35,14.032,9.217,4.35,9.165,-0.517,4.297)
a
=
#(90,90,30,-30,-20,20,90,90,90,90,90,90,90,90,30,30,-30,-30,30,30,-30,-30,-20,-20,20,20,-20,-20,20,20,75,50,50,75,50,50,-30,-30,80,80,60,60,60,60,40,70
,-55,55,40,70,-55,55,71.5,71.5,0,-71.5,71.5,71.5,0,-71.5)
b
=
#(45,-45,0,0,0,0,-5,5,95,-95,65,-65,-15,15,45,-45,45,-45,120,-120,120,-120,30,-30,30,-30,90,-90,90,-90,0,75,-75,0,75,-75,0,-45,-45,-45,30,30,235,235,-120
,60,-40,-18,-120,60,-40,-18,-60,60,0,0,-60,60,0,0)
ri = #(1,1,1,0,1,1,1,1,1,1,1,1,1,1,1,1,1,0,1,0,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,0,1,1,1,1,1,0,1,1,0,1,1,1,1,1,1,0,1,0,1,1)
for l = 1 to 60 do(
if ri[l] > 0 then (r[l] = torus radius1:4.8 radius2:0.4 segs:20 sides:12 position: [x[l],y[l],z[l]] wirecolor: [200,200,0]
rotate r[l]
a[l] [1,0,0]
rotate r[l] b[l] [0,0,1]
))
select #(r[1],r[2],r[3],r[5],r[6],r[7],r[8],r[9],r[10],r[11],r[12],r[13],r[14],r[15],r[16],r[17],r[19]
,r[21],r[22],r[23],r[24],r[25],r[26],r[27],r[28],r[29],r[30],r[31],r[32],r[33],r[34],r[35],r[36],r[37],r[38],r[39]
,r[41],r[42],r[43],r[44],r[45],r[47],r[48],r[50],r[51],r[52],r[53],r[54],r[55],r[57],r[59],r[60])
macros.run "Modifier stack" "convert_to_Mesh"
element1 = copy r[1] wirecolor: [200,200,0]
for
ll = 2 to 60 do(if ri[ll] > 0 then (attach element1 r[ll]))
delete r[1]
element1.material = newmat[12]
--
adenosine, cytosine, guanine, timidine, uracil, inosine, pseudouracil,
dihydrouracil, methyl inosine, x-circles maximum, w-methyl-2-guanine,
v-methyl-1-guanine
if (seq[k] == nucl[10]) then (ai =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); axi = #(1,1,1,1,1,1,1,1); ayi =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); nuclcol = nuclcolor[10]; nc=10)
if
(seq[k] == nucl[1]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol =
nuclcolor[1]; nc=1)
if (seq[k] == nucl[2]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[2]; nc=2)
if
(seq[k] == nucl[3]) then (ai = #(0,1,0,1,1,1,1,1,1,1,1,1,0,1,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,0,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol =
nuclcolor[3]; nc=3)
if (seq[k] == nucl[4]) then (ai =
#(0,1,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,0,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[4]; nc=4)
if
(seq[k] == nucl[5]) then (ai = #(0,1,0,1,1,1,1,1,1,1,0,1,0,0,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,0,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol =
nuclcolor[5]; nc=5)
if (seq[k] == nucl[6]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol = nuclcolor[6]; nc=6)
if
(seq[k] == nucl[7]) then (ai = #(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol =
nuclcolor[7]; nc=7)
if (seq[k] == nucl[8]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[8]; nc=8)
if
(seq[k] == nucl[9]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol =
nuclcolor[9]; nc=9)
--
element0 = torus radius1:0.1 radius2:0.05 segs:3 sides:3 position:[0,0,0] wirecolor: nuclcol
Converttomesh element0
for i = 1 to 3 do for j = 1 to 5 do (n = i + 3*(j-1)
if ai[n] > 0 then (anuk[n] = sphere radius:0.5 segs:20 position:[10*(i-2),10*(j-5),0] wirecolor: [0,0,0]
Converttomesh anuk[n]
anuk[n].material = newmat[14]
attach element0 anuk[n]))
--
for i = 1 to 3 do for j = 1 to 5 do (n = i + 3*(j-1)
if
ai[n] > 0 then (aa[n] = torus radius1:4.8 radius2:0.4 segs:20
sides:12 position:[10*(i-2),10*(j-5),10*0.5] wirecolor: nuclcol
aa[n+12]=copy
aa[n] wirecolor: nuclcol; move aa[n] [0,0,-10]; select
#(aa[n],aa[n+12]); macros.run "Modifier stack" "convert_to_Mesh"
attach element0 aa[n];attach element0 aa[n+12]))
for i = 1 to 2 do for j = 1 to 4 do (m = i + 2*(j-1)
if
axi[m] > 0 then (ax[m] = torus radius1:4.8 radius2:0.4 segs:20
sides:12 position:[10*(3*(i-1.5)),10*(j-4),0] wirecolor: nuclcol
rotate ax[m] 90 [0,1,0]; Converttomesh ax[m]
attach element0 ax[m]))
for i = 1 to 3 do for j = 1 to 5 do (m = i + 3*(j-1)
if
ayi[m] > 0 then (ay[m] = torus radius1:4.8 radius2:0.4 segs:20
sides:12 position:[10*(i-2),10*(j-4.5),0] wirecolor: nuclcol
rotate ay[m] 90 [1,0,0]; Converttomesh ay[m]
attach element0 ay[m])
element0.material = newmat[nc])
--
if ci[k] > 0 then (
--
if
(complement[k] == nucl[10]) then (ai =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); axi = #(1,1,1,1,1,1,1,1); ayi =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); nuclcol = nuclcolor[10]; nc=10)
if
(complement[k] == nucl[1]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0);
nuclcol = nuclcolor[1]; nc=1)
if (complement[k] == nucl[2]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[2]; nc=2)
if
(complement[k] == nucl[3]) then (ai = #(0,1,0,1,1,1,1,1,1,1,1,1,0,1,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,0,0,0,0,0,0,0,0,1,0,1,0,1,0);
nuclcol = nuclcolor[3]; nc=3)
if (complement[k] == nucl[4]) then (ai =
#(0,1,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,0,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[4]; nc=4)
if
(complement[k] == nucl[5]) then (ai = #(0,1,0,1,1,1,1,1,1,1,0,1,0,0,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,0,0,0,0,0,0,1,0,1,0,1,0,0,0);
nuclcol = nuclcolor[5]; nc=5)
if (complement[k] == nucl[6]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol = nuclcolor[6]; nc=6)
if
(complement[k] == nucl[7]) then (ai = #(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0);
nuclcol = nuclcolor[7]; nc=7)
if (complement[k] == nucl[8]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[8]; nc=8)
if
(complement[k] == nucl[9]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0);
nuclcol = nuclcolor[9]; nc=9)
element4 = box length:0.1 width:0.1 height:0.1 position:[0,0,-0.05] wirecolor: nuclcol
Converttomesh element4
--
for i = 1 to 3 do for j = 1 to 5 do (n = i + 3*(j-1)
if ai[n] > 0 then (anuk[n] = sphere radius:0.5 segs:20 position:[10*(i-2),10*(j-5),0] wirecolor: [0,0,0]
Converttomesh anuk[n]
anuk[n].material = newmat[14]
attach element4 anuk[n]))
--
for i = 1 to 3 do for j = 1 to 5 do (n = i + 3*(j-1)
if
ai[n] > 0 then (aa[n] = torus radius1:4.8 radius2:0.4 segs:20
sides:12 position:[10*(i-2),10*(j-5),10*0.5] wirecolor: nuclcol
aa[n+12]=copy
aa[n] wirecolor: nuclcol; move aa[n] [0,0,-10]; select
#(aa[n],aa[n+12]); macros.run "Modifier stack" "convert_to_Mesh"
attach element4 aa[n];attach element4 aa[n+12]))
for i = 1 to 2 do for j = 1 to 4 do (m = i + 2*(j-1)
if
axi[m] > 0 then (ax[m] = torus radius1:4.8 radius2:0.4 segs:20
sides:12 position:[10*(3*(i-1.5)),10*(j-4),0] wirecolor: nuclcol
rotate ax[m] 90 [0,1,0]; Converttomesh ax[m]
attach element4 ax[m]))
for i = 1 to 3 do for j = 1 to 5 do (m = i + 3*(j-1)
if
ayi[m] > 0 then (ay[m] = torus radius1:4.8 radius2:0.4 segs:20
sides:12 position:[10*(i-2),10*(j-4.5),0] wirecolor: nuclcol
rotate ay[m] 90 [1,0,0]; Converttomesh ay[m]
attach element4 ay[m])
element4.material = newmat[nc])
--
element5 = copy element2
element6 = copy element1
attach element4 element6
attach element4 element5
element4.pivot = [0,0,0]
if ci[k] == 1 then (rotate element4 180 [1,0,0])
if ci[k] == 2 then (rotate element4 180 [1,0,0]; rotate element4 -90 [0,1,0]; move element4 [0,-20,0])
if ci[k] ==
3 then (rotate element4 180 [1,0,0]; rotate element4 180 [0,1,0])
if ci[k] == 4 then (rotate element4 180 [1,0,0]; rotate element4 22.5 [0,1,0]; move element4 [0,10,0])
if
ci[k] == 5 then (rotate element4 180 [1,0,0]; rotate element4 45
[0,1,0]; rotate element4 45 [0,0,1]; move element4 [-20,20,0])
if ci[k] == 6 then (rotate element4 180 [1,0,0]; rotate element4 -135 [0,1,0])
attach element1 element4)
-- H
h1 = sphere radius:0.4 segs:20 position:[0,10,0] wirecolor: [255,255,255]
h0 = sphere radius:0.4 segs:20 position:[0,-10,0] wirecolor: [255,255,255]
h2 = sphere radius:0.4 segs:20 position:[10,0,0] wirecolor: [255,255,255]
h3 = sphere radius:0.4 segs:20 position:[-10,0,0] wirecolor: [255,255,255]
Converttomesh
h1
Converttomesh h0
Converttomesh h2
Converttomesh h3
attach h1 h2
attach h1 h3
attach h1 h0
h1.material = newmat[13]
attach element1 h1
-- first vector
gr1 = group #(element0, element1,trnk)
gr1.pivot = [-37.451, -2.887, 14.138]
rotate gr1 anglecomp1[k] [0, -27.384, -9.84]
ungroup gr1
-- second vector
gr2 = #(element0, element1, element2,trnk)
gr2.pivot = [-43.391, -10.855, 7.631]
rotate gr2 anglecomp2[k] [-11.880, -2.245,
-8.094]
ungroup gr2
-- translation vector
attach trnk element0
attach trnk element1
attach trnk element2
move trnk [5.511 ,-49.39, -25.725]
trnk.pivot = [-26, -58, -10]
-- translation angles
rotate trnk 50 [0, 1, 0]
rotate trnk 35 [0, 0, 1])
Для создания пикотехнологических моделей протяжённых участков ДНК/РНК созданы упрощённые (многогранные) модели нуклеотидов...
Теперь скрипт показывает водородные связи белыми кубиками (только для мажоров).
Скрипт, который строит упрощённую (многогранную) пикотехнологическую модель ДНК/РНК:
-- Nanoworld Laboratory
-- Alexander Kushelev
-- Pikotechnological
DNA / RNA - model
-- http://nanoworld.narod.ru/
aa = #(); ax = #(); ay = #(); nuclcol = #()
--
adenosine, cytosine, guanine, timidine, uracil, inosine, pseudouracil,
dihydrouracil, methyl inosine, x-circles maximum, w-methyl-2-guanine,
v-methyl-1-guanine
nucl = #("a","c","g","t","u","i","p","d","m","x","w","v")
nuclcolor
=
#([200,0,0],[200,100,100],[0,200,200],[0,0,200],[100,0,200],[200,0,200],[150,0,0],[100,50,0],[0,80,50],[100,100,100])
--*****************************************************************************************************************
seq = #("a","c","c","a","c","c","u","g","c","u"
-- 11
,"c","a","g","g","c","c","u","u","a","g"
-- 21
,"c","p","t","g","g","c","c","u","c","d"
-- 31
,"g","g","a","g","a","g","g","g","p","m"
-- 41
,"c","g","i","u","u","c","c","c","u","c"
-- 51
,"w","c","g","c","g","a","d","g","g","c"
-- 61
,"d","g","a","u","g","c","g","v","u","g"
-- 71 76
,"u","g","c","g","g","g")
--*****************************************************************************************************************
-- seq = #("c","c","c","c","c","c","c","c","c","c","c","c","c","c","c","c","c","c","c","c")
-- seq = #("i","i","i","i","i","i","i","i","i","i","i","i","i","i","i","i","i","i","i")
--*****************************************************************************************************************
-- complement
complement = #("0","0","0","0","g","g","g","c","g","u"
-- 11
,"g","u","c","c","g","g","0","t","p","0"
-- 21
,"0","0","0","0","0","0","0","0","0","w"
-- 31
,"0","0","0","c","u","c","c","c","i","0"
-- 41
,"0","0","0","0","0","0","0","0","0","0"
-- 51
,"0","g","c","g","u","a","g","d","0","0"
-- 61
,"0","0","0","0","0","0","0","0","0","0"
-- 71 76
,"0","0","0","0","0","0")
-- complement = #("t","t","t","t","t","t","t","t","t","t","t","t","t","t","t","t","t","t","t","t")
-- complement = #("p","p","p","p","p","p","p","p","p","p","p","p","p","p","p","p","p","p","p","p")
ci = #(0,0,0,0,1,1,1,1,1,1
-- 11
,1,1,1,1,1,1,0,2,3,0
-- 21
,0,0,0,0,0,0,0,0,0,0
-- 31
,0,0,0,1,1,1,1,1,3,0
-- 41
,0,0,0,0,0,0,0,0,0,0
-- 51
,0,1,1,1,4,5,6,6,0,0
-- 61
,0,0,0,0,0,0,0,0,0,0
-- 71 76
,0,0,0,0,0,0)
-- ci = #(2,2,2,2,2,2,2,2,2,2,2,2,2,2,2,2,2,2,2,2)
-- ci = #(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1,1)
-- ci = #(3,3,3,3,3,3,3,3,3,3,3,3,3,3,3,3,3,3,3,3)
--*****************************************************************************************************************
--
turn
anglecomp1 = #(0,0,0,0,0,0,0,0,0,0
-- 11
,
0, 0, 0, 0,
0, 0, 0, 120, 0, 0
-- 21
,-120,
0, 0, 0, 0,
0, 0, 0, 0, 0
-- 31
, 0, 0, 0, 0, 0,
0, 0, -20, -40, 40
-- 41
, 0,
-30, 0, 0, 0, 0,
0, 0, 0, 0
-- 51
,
0, 0, 0, 0,
0, 0, 0, 0, 120, 0
-- 61
,
0, 0, 0, 0,
0, 0, 0, 0,
0,
0
-- 71 76
, 0, 0, 0, 0, 0, 0)
anglecomp2 = #(0,0,0,0,0,0,0,0,0,0
-- 11
, 120, 0, 0, 0, 0, 0, 0, 0, -30, -60
-- 21
,
120, 0, 0, 0, 0,
0, 0, 0, 0,
0
-- 31
, 0, 0, 0,
0, 0, 0, 0,
0, 0,-120
-- 41
, 0, 120,
0, 0, 0, 0,
0, 0, 0, 0
-- 51
,
0, 0, 0, 0,
0, 0, 0, 0, 60,
0
-- 61
,0 ,
0, 0, 0, 0, 0, 0, 0, 0, 0
-- 71 76
, 0, 0, 0, 0, 0, 0)
--*****************************************************************************************************************
trnk = torus radius1:0.1 radius2:0.04 segs:3 sides:3 position: [0,0,0]
wirecolor:[200,200,200]
Converttomesh trnk
newmat = multimaterial name:"MyMultiMat" numsubs: (14)
newmat[1].faceted = on
newmat[2].faceted = on
newmat[3].faceted = on
newmat[4].faceted = on
newmat[5].faceted = on
newmat[6].faceted = on
newmat[7].faceted = on
newmat[8].faceted = on
newmat[9].faceted = on
newmat[10].faceted = on
newmat[11].faceted = on
newmat[12].faceted = on
newmat[13].faceted = on
newmat[14].faceted
= on
newmat[1].diffuse = (color 200 0 0)
newmat[2].diffuse = (color 200 100 100)
newmat[3].diffuse = (color 0 200 200)
newmat[4].diffuse = (color 0 0 200)
newmat[5].diffuse = (color 100 0 200)
newmat[6].diffuse = (color 200 0 200)
newmat[7].diffuse = (color 150 0 0)
newmat[8].diffuse = (color 100 50 0)
newmat[9].diffuse = (color 0 80 50)
newmat[10].diffuse = (color 100 100 100)
newmat[11].diffuse = (color 0 200 0)
newmat[12].diffuse = (color 200 200
0)
newmat[13].diffuse = (color 255 255 255)
newmat[14].diffuse = (color 0 0 0)
--
m3 = mesh vertices: #([-2.828,-45.5,-2.828]
,[2.828,-45.5,-2.828],[2.828,-45.5,2.828],[-2.828,-45.5,2.828],[-2.828,-61.5,-2.828],[2.828,-61.5,-2.828]
,[2.828,-61.5,2.828],[-2.828,-61.5,2.828],[-2.828,-53.5,-8.485],[-5.657,-49.5,-5.657],[-8.485,-53.5,-2.828]
,[-5.657,-57.5,-5.657],[8.485,-53.5,2.828],[5.657,-49.5,5.657],[2.828,-53.5,8.485],[5.657,-57.5,5.657]
,[5.657,-49.5,-5.657],[2.828,-53.5,-8.485],[5.657,-57.5,-5.657],[8.485,-53.5,-2.828],[-5.657,-49.5,5.657]
,[-8.485,-53.5,2.828],[-5.657,-57.5,5.657],[-2.828,-53.5,8.485]) \
faces: #([1,3,2],[1,4,3],[5,6,7],[5,7,8],[9,11,10],[9,12,11],[13,14,15],[13,15,16],[17,19,18],[17,20,19],[21,22,23],[21,23,24]
,[1,2,17],[17,18,9],[9,10,1],[1,17,9],[10,22,4],[4,1,10],[10,11,22],[22,21,4],[11,5,23],[23,22,11],[11,12,5],[5,8,23]
,[12,18,6],[6,5,12],[12,9,18],[18,19,6],[8,16,24],[24,23,8],[16,15,24],[8,7,16],[15,3,21],[21,24,15],[15,14,3]
,[3,4,21],[14,20,2],[2,3,14],[14,13,20],[20,17,2],[13,7,19],[19,20,13],[13,16,7],[7,6,19])
\
wirecolor: [255,255,0]
m3.material = newmat[12]
m4 = copy m3 wirecolor: [255,255,0]
m4.material = newmat[12]
m4.pivot = [0,-40,0]
rotate m4 -120 [0,0,1]
move m4 [-7.5,-0.5,0]
m6 = copy m3 wirecolor: [255,255,0]
m6.material = newmat[12]
m13 = copy m3 wirecolor: [255,255,0]
m13.material = newmat[12]
m13.pivot = [0,-66,0]
rotate m13 -72 [1,0,0]
m14 = copy m13 wirecolor: [0,255,0]
m14.material = newmat[11]
g1 = group #(m6, m14)
g1.pivot = [0,-40,0]
rotate g1 -45 [0,0,1]
move g1 [-5.5,2.5,0]
ungroup g1
m5 = copy m6 wirecolor: [255,255,0]
m5.material = newmat[12]
m7 = copy m4 wirecolor: [255,255,0]
m7.material = newmat[12]
g2 = group #(m5, m7)
g2.pivot = [0,-40,0]
rotate g2 180 [0,1,0]
ungroup g2
m8 = mesh vertices: #([-4,0,-8],[0,-4,-8],[4,0,-8],[0,4,-8],[-4,0,8],[0,-4,8],[4,0,8],[0,4,8]
,[-8,-4,0],[-8,0,-4],[-8,4,0],[-8,0,4],[8,-4,0],[8,0,-4],[8,4,0],[8,0,4]
,[0,-8,-4],[-4,-8,0],[0,-8,4],[4,-8,0],[0,8,-4],[-4,8,0],[0,8,4],[4,8,0])
\
faces: #([1,3,2],[1,4,3],[5,6,7],[5,7,8],[9,11,10],[9,12,11],[13,14,15],[13,15,16],[17,19,18],[17,20,19],[21,22,23],[21,23,24]
,[1,2,17],[17,18,9],[9,10,1],[1,17,9],[10,22,4],[4,1,10],[10,11,22],[22,21,4],[11,5,23],[23,22,11],[11,12,5],[5,8,23]
,[12,18,6],[6,5,12],[12,9,18],[18,19,6],[8,16,24],[24,23,8],[16,15,24],[8,7,16],[15,3,21],[21,24,15],[15,14,3]
,[3,4,21],[14,20,2],[2,3,14],[14,13,20],[20,17,2],[13,7,19],[19,20,13],[13,16,7],[7,6,19]) \
wirecolor: [0,255,0]
m8.material
= newmat[11]
rotate m8 45 [1,0,0]
rotate m8 90 [0,0,1]
move m8 [-37.5,0,0]
m9 = copy m8 wirecolor: [0,255,0]
m9.material = newmat[11]
m9.pivot = [-37.5,-13,-0.5]
rotate m9 72 [1,0,0]
m10 = copy m8 wirecolor: [0,255,0]
m10.material = newmat[11]
m10.pivot = [-37.5,-13,-0.5]
rotate m10 144 [1,0,0]
m11 = copy m8 wirecolor: [0,255,0]
m11.material = newmat[11]
m11.pivot = [-37.5,-13,-0.5]
rotate m11 18 [1,0,0]
rotate m11 90 [0,0,1]
rotate m11 180 [1,0,0]
rotate m11 -18 [1,0,0]
move m11 [0,7,24]
m12 = copy m9 wirecolor: [255,255,0]
m12.material = newmat[12]
m12.pivot = [-37.5,-23.5,15]
rotate m12 -120 [0,0,1]
attach m3 m4
attach m5 m7
attach m6 m13
attach m3 m5
attach m3 m6
attach m8 m9
attach m8 m11
attach m3 m10
attach m12 m14
attach m3 m12
--
for k = 52 to 60 do(
--
adenosine, cytosine, guanine, timidine, uracil, inosine, pseudouracil,
dihydrouracil, methyl inosine, x-circles maximum, w-methyl-2-guanine,
v-methyl-1-guanine
if (seq[k] == nucl[10]) then (ai =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); axi = #(1,1,1,1,1,1,1,1); ayi =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); nuclcol = nuclcolor[10]; nc=10)
if
(seq[k] == nucl[1]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol =
nuclcolor[1]; nc=1)
if (seq[k] == nucl[2]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[2]; nc=2)
if
(seq[k] == nucl[3]) then (ai = #(0,1,0,1,1,1,1,1,1,1,1,1,0,1,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,0,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol =
nuclcolor[3]; nc=3)
if (seq[k] == nucl[4]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[4]; nc=4)
if
(seq[k] == nucl[5]) then (ai = #(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol =
nuclcolor[5]; nc=5)
if (seq[k] == nucl[6]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol = nuclcolor[6]; nc=6)
if
(seq[k] == nucl[7]) then (ai = #(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol =
nuclcolor[7]; nc=7)
if (seq[k] == nucl[8]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[8]; nc=8)
if
(seq[k] == nucl[9]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi =
#(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol =
nuclcolor[9]; nc=9)
--
element1 = box length:1 width:1 height:1 position:[0,0,-0.5] wirecolor: nuclcol
Converttomesh element1
for i = 1 to 3 do for j = 1 to 5 do (n = i + 3*(j-1)
if ai[n] > 0 then (aa[n] = box length:10 width:10 height:10 position:[10*(i-2),10*(j-5),10*0.5-10] wirecolor: nuclcol
select #(aa[n]); macros.run "Modifier stack" "convert_to_Mesh"
attach element1 aa[n]
element1.material = newmat[nc]))
element2 = copy m8
element3 = copy m3
attach element1 element3
--
if ci[k] > 0 then (
--
if
(complement[k] == nucl[10]) then (ai =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); axi = #(1,1,1,1,1,1,1,1); ayi =
#(1,1,1,1,1,1,1,1,1,1,1,1,1,1,1); nuclcol = nuclcolor[10]; nc=10)
if
(complement[k] == nucl[1]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0);
nuclcol = nuclcolor[1]; nc=1)
if (complement[k] == nucl[2]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[2]; nc=2)
if
(complement[k] == nucl[3]) then (ai = #(0,1,0,1,1,1,1,1,1,1,1,1,0,1,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,0,0,0,0,0,0,0,0,1,0,1,0,1,0);
nuclcol = nuclcolor[3]; nc=3)
if (complement[k] == nucl[4]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[4]; nc=4)
if
(complement[k] == nucl[5]) then (ai = #(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0);
nuclcol = nuclcolor[5]; nc=5)
if (complement[k] == nucl[6]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0); nuclcol = nuclcolor[6]; nc=6)
if
(complement[k] == nucl[7]) then (ai = #(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0);
nuclcol = nuclcolor[7]; nc=7)
if (complement[k] == nucl[8]) then (ai =
#(0,0,0,1,1,1,1,1,1,1,0,1,0,0,0); axi = #(0,0,1,1,0,0,0,0); ayi =
#(0,1,0,0,0,0,0,1,0,1,0,1,0,0,0); nuclcol = nuclcolor[8]; nc=8)
if
(complement[k] == nucl[9]) then (ai = #(0,0,0,1,1,1,1,1,1,1,1,1,0,1,0);
axi = #(0,0,1,1,0,0,0,0); ayi = #(0,1,0,0,0,0,0,0,0,1,0,1,0,1,0);
nuclcol = nuclcolor[9]; nc=9)
element4 = box length:1 width:1 height:1 position:[0,0,-0.5] wirecolor: nuclcol
Converttomesh element4
for i = 1 to 3 do for j = 1 to 5 do (n = i + 3*(j-1)
if ai[n] > 0 then (aa[n] = box length:10 width:10 height:10 position:[10*(i-2),10*(j-5),10*0.5-10] wirecolor: nuclcol
select #(aa[n]); macros.run "Modifier stack" "convert_to_Mesh"
attach element4 aa[n]
element4.material = newmat[nc]))
element5 = copy
m8
element6 = copy m3
attach element4 element6
attach element4 element5
element4.pivot = [0,0,0]
if ci[k] == 1 then (rotate element4 180 [1,0,0])
if ci[k] == 2 then (rotate element4 180 [1,0,0]; rotate element4 -90 [0,1,0]; move element4 [0,-20,0])
if ci[k] == 3 then (rotate element4 180 [1,0,0]; rotate element4 180 [0,1,0])
if ci[k] == 4 then (rotate element4 180 [1,0,0]; rotate element4 22.5 [0,1,0]; move element4 [0,10,0])
if
ci[k] == 5 then (rotate element4 180 [1,0,0]; rotate element4 45
[0,1,0]; rotate element4 45 [0,0,1]; move element4 [-20,20,0])
if ci[k] == 6 then (rotate element4 180 [1,0,0]; rotate element4 -135 [0,1,0])
attach element1 element4)
--
h1 = box length:9.9 width:9.9 height:9.9 position:[0,10,-4.95] wirecolor: [255,255,255]
h0 = box length:9.9 width:9.9 height:9.9 position:[0,-10,-4.95] wirecolor: [255,255,255]
h2 = box length:9.9 width:9.9 height:9.9 position:[10,0,-4.95] wirecolor: [255,255,255]
h3 = box length:9.9 width:9.9
height:9.9 position:[-10,0,-4.95] wirecolor: [255,255,255]
Converttomesh h1
Converttomesh h0
Converttomesh h2
Converttomesh h3
attach h1 h2
attach h1 h3
attach h1 h0
h1.material = newmat[13]
attach element1 h1
-- first vector
gr1 = group #(element1,trnk)
gr1.pivot = [-37.451, -2.887, 14.138]
rotate gr1 anglecomp1[k] [0, -27.384, -9.84]
if k == 40 then (rotate gr1 -90 [0, 0, 1] ; rotate gr1 -20 [0, 1, 0] )
ungroup gr1
-- second
vector
gr2 = #(element1, element2, trnk)
gr2.pivot = [-43.391, -10.855, 7.631]
rotate gr2 anglecomp2[k] [-11.880, -2.245, -8.094]
ungroup gr2
-- translation vector
attach trnk element1
attach trnk element2
move trnk [0,-50, 0]
trnk.pivot = [-60,0,0]
-- translation angles
rotate trnk 36 [0, 1, 0]
-- rotate trnk 35 [0, 0, 1])
--
delete m3
delete m8
trnk.rotation.controller[2].controller.value = 0
animate on
at time 100 trnk.rotation.controller[2].controller.value
= 360
Всё это, дорогие читатели, Вы можете читать благодаря постраничной копии форума лаборатории Наномир, уничтоженного преступной группировкой, совершившей (и продолжающей совершать) сотни преступлений против лаборатории Наномир с 2006 года по настоящее время.
Добро пожаловать на новый форум лаборатории Наномир!